生物信息学6

第三章 序列分析与联配
序列分析是生物信息学最主要的研究内容之一,它可以分为两个主要部分:一是序列组成(特别是涉及到基因组层次上)分析,二是序列之间的比较分析。两条序列或多条序列间的比对或联配(alignment)的目的,是对它们的序列相似性进行评估,找出这些序列中结构或功能相似性区域等。通过联配未知序列与已知序列(其功能或结构等已知)的相似程度,我们可以判断或推测未知序列的结构与功能。
第一节 序列组成及单一序列分析1
一. 碱基组成
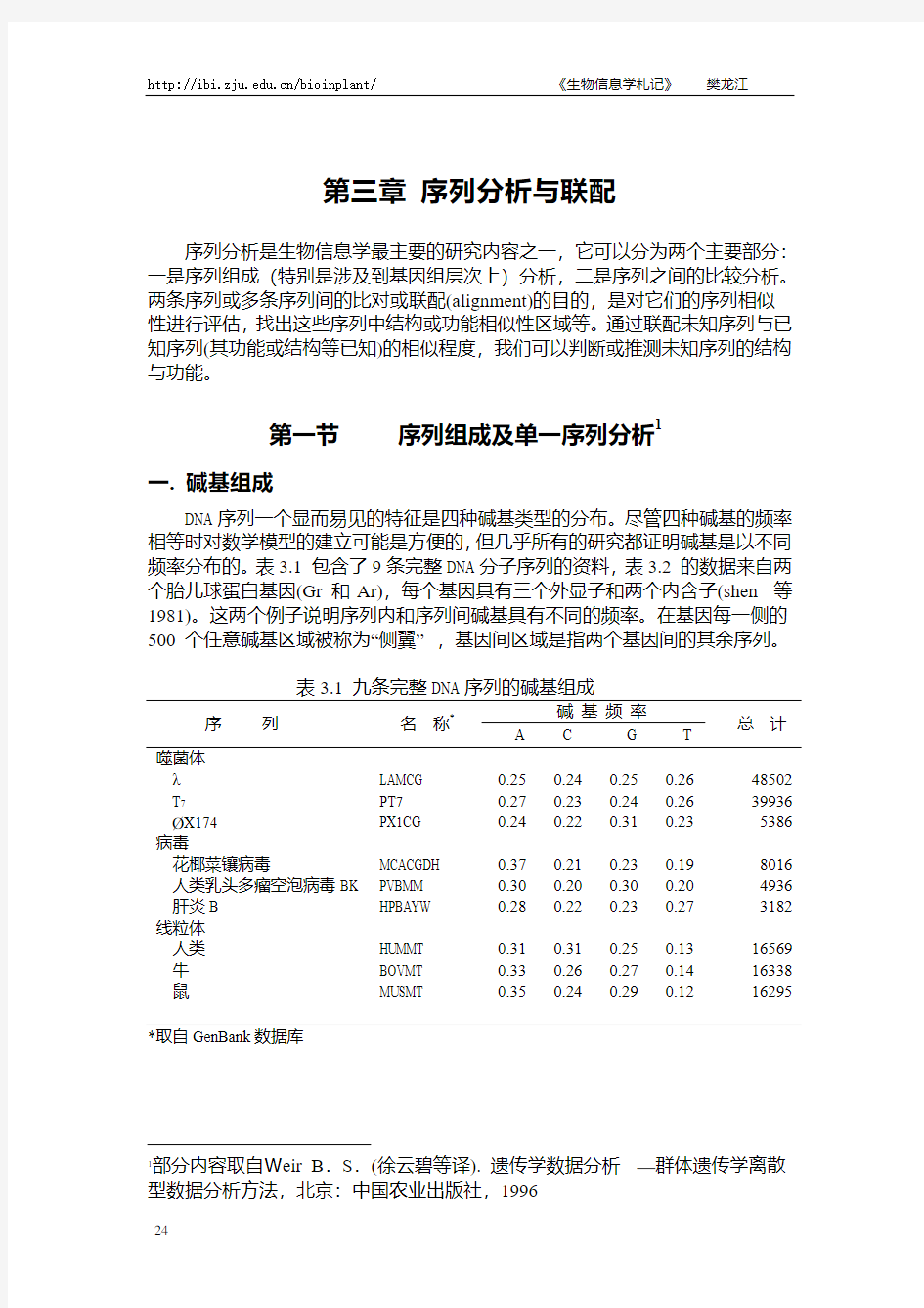
DNA序列一个显而易见的特征是四种碱基类型的分布。尽管四种碱基的频率相等时对数学模型的建立可能是方便的,但几乎所有的研究都证明碱基是以不同频率分布的。表3.1包含了9条完整DNA分子序列的资料,表3.2的数据来自两个胎儿球蛋白基因(Gr和Ar),每个基因具有三个外显子和两个内含子(shen等1981)。这两个例子说明序列内和序列间碱基具有不同的频率。在基因每一侧的500 个任意碱基区域被称为“侧翼”,基因间区域是指两个基因间的其余序列。
表3.1九条完整DNA序列的碱基组成
碱 基 频 率
序 列 名 称*
A C G T
总 计噬菌体
λ
T7
?X174
病毒
花椰菜镶病毒
人类乳头多瘤空泡病毒BK 肝炎B
线粒体
人类
牛
鼠 LAMCG
PT7
PX1CG
MCACGDH
PVBMM
HPBAYW
HUMMT
BOVMT
MUSMT
0.25
0.27
0.24
0.37
0.30
0.28
0.31
0.33
0.35
0.24
0.23
0.22
0.21
0.20
0.22
0.31
0.26
0.24
0.25
0.24
0.31
0.23
0.30
0.23
0.25
0.27
0.29
0.26
0.26
0.23
0.19
0.20
0.27
0.13
0.14
0.12
48502
39936
5386
8016
4936
3182
16569
16338
16295
*取自GenBank数据库
1部分内容取自Weir B.S.(徐云碧等译). 遗传学数据分析—群体遗传学离散型数据分析方法,北京:中国农业出版社,1996
表3.2 人类胎儿球蛋白基因不同区段的碱基组成
区 段
长 度 A C G T 5′例翼(2) 3′例翼(2) 内含子(4) 外显子(6) 基因间(1)
1000 1000 1996 882 2487
0.33 0.29 0.27 0.24 0.32
0.23 0.15 0.17 0.25 0.19
0.22 0.26 0.27 0.28 0.18
0.22 0.30 0.29 0.22 0.31
*数据来自EMBL 数据库HSGLBN 基因
二.碱基相邻频率
分析DNA 序列的主要困难之一是碱基相邻的频率不是独立的。碱基相邻的频率一般不等于单个碱基频率的乘积:如果P u 是序列中碱基u 的频率,且P uv 为两个相邻碱基u 和v 的频率,则
v u uv p p p ≠ Nussinov(1984)研究了两碱基相邻的频率(表3.3)。数据来自166个脊椎动物的DNA 序列,总长136731个碱基。表中的比值为16种二个碱基相邻的频率除以相应的单个碱基频率的乘积。
表3.3 脊椎动物中两碱基的相邻频率
相邻碱基对 观测频率/期望频率*
TG
CT CC AG AA CA GG TT GA TC GC AT AC GT TA CG
1.29 1.26 1.18 1.16 1.15 1.15 1.14 1.07 10.4 1.00 0.99 0.85 0.84 0.82 0.65 0.42
*期望频率为相应两个单个碱基频率的乘积
作为一个特别的例子,图3.1给出了鸡血红蛋白β链的mRNA 编码区的438个碱基。表3.4列出了4种碱基和16种两碱基的数目。将该表看作4×4的表,
计算行列独立性的卡方统计量,得到,表明行(第一碱基)
列(第二碱基)之间存在明显的关联。
)92.16(3.592
9,05.02==χχ
GTGCACTGGA CTGCTGAGGA GAAGCAGCTC ATCACCGGCC TCTGGGCAA GGTCAATGTG 60 GCCGAATGTG GGGCCGAAGC CCTGGCCAGG CTGCTGATCG TCTACCCCTG GACCCAGAGG 120 TTCTTTGCGT CCTTTGGGAA CCTCTCCAGC CCCACTGCCA TCCTTGGCAA CCCCATGGTC 180 CGCGCCCACG GCAAGAAAGT GCTCACCTCC TTTGGGGATG CTGTGAAGAA CCTGGACAAC 240 ATCAAGAACA CCTTCTCCCA
ACTGTCCGAA
CTGCATTGTG ACAAGCTGCA TGTGGACCCC 300
GAGAACTTCA GGCTCCTGGG TGACATCCTC ATCATTGTCC TGGCCGCCCA CTTCAGCAAG 360 GACTTCACTC CTGAATGCCA GGCTGCCTGG CAGAAGCTGG TCCGCGTGGT GGCCCATGCC 420 CTGGCTCGCA AGTACCAC
图3.1 鸡β球蛋白基因编码区的DNA 序列 (GenBank:CHKHBBM,记录号J00860)
表3.4 图3.1鸡β球蛋白基因序列的相邻碱基分布
第二碱基
A C G T 总计 第一碱基 A 23 26 23 15 87 C 37 51 14 41 143 G 25 38 36 19 118 T 2 29 41 14 89 总计 87 144 117 89 437
在编码区,存在某种约束来限制DNA 序列编码氨基酸。在密码子水平上,这一约束与碱基相邻频率有关。表3.5列出了遗传密码和图3.1序列中各密码子数量。尽管数目很小,难以作出有力的统计结论,但编码同一氨基酸的不同密码子(同义密码子)好像不是等同存在的。这种密码子偏倚必定与两碱基相邻频率水平有关。表3.5还清楚地表明,由于密码子第3位置上碱基的改变常常不会改变氨基酸的类型,因而对第3位置上碱基的约束要比第 2位碱基小得多。
表3.5 64种可能的碱基三联体密码子及相应的氨基酸数(据图3.1序列)
UUU Phe 3 UUC Phe 5 UUA Leu 0 UUG Leu 0
CUU Leu 1 CUC Leu 6 CUA Leu 0 CUG Leu 11
AUU Ile 1 AUC Ile 6 AUA Ile 0 AUG Met 1
GUU Val 0 GUC Val 5 GUA Val 0 GUG Val7 UCU Ser 0 UCC Ser 5 UCA Ser 0 UCG Ser 0
CCU Pro 1 CCC Pro 4 CCA Pro 0 CCG Pro 0
ACU Thr 3 ACC Thr 4 ACA Thr 0 ACG Thr 0
GCU Ala 4 GCC Ala 11 GCA Ala 0 GCG Ala 1
UAU Tyr 0 UAC Tyr 2 UAA Stop 0 UAG Stop 0
CAU His 3 CAC His 4 CAA Gln 1 CAG Gln 0
AAU Asn 1 AAC Asn 6 AAA Lys 1 AAG Lys 9
GAU Asp 1 GAC Asp 5 GAA Glu 4 GAG Glu 3
UGU Cys 2 UGC Cys 1 UGA Stop 0 UGG Trp 4
CGU Arg 0 CGC Arg 3 CGA Arg 0 CGG Arg 0
AGU Sre 0 AGC Ser 2 AGA Arg 0 AGG Arg 3
GGU Gly 1 GGC Gly 4 GGA Gly o GGG Gly 3
相邻碱基之间的关联将导致更远碱基之间的关联,这些关联延伸距离的估计可以从马尔科夫链(Markov chain)理论得到(Javare 和Giddings,1989)。在不援引任何生物学机制的情况下,第k 阶马尔科夫链假定在序列中某一位置上碱基的存在只取决于前面k 个位置上的碱基。一阶链假定一个特定碱基存在于位置i 的概率只取取决于在位置i-1的4种碱基概率。相互独立的碱基所组成的序列将与0阶马尔科夫链相对应。阶可以通过似然法估计。同时,马尔科夫链分析更适应于基因组水平,而非单一序列(基因)。相关内容可参见第四章第2节。
三.同向重复序列分析
除了分析整个序列碱基关联程度的特征外,我们常对寻找同向重复序列(direct repeats)之类的问题感兴趣。Karlin等(1983)给出了完成这一分析的有效算法。该法采用由特定的几组碱基字母组成的不同亚序列或称为字码(word)。只需要对整个序列搜索一次。给一碱基赋以值α,例如A、C、G、T的值为0、1、2、3。由X 1、X 2、…、X k 共k 个字母组成的每一种不同的字码按计算
字码值。这些值的取值范围为1到4∑=?+k
i i k i 141αk 。
例如,5字码TGACC的值为1+3×44+2×43
+0×42+1×41+1×40
=459。可先从低k值的字码开始搜索。记录序列中每一个位置k 字码的字码值。只有在发现k字码长度重复的那些位置考虑进行长度大于k的字码搜索。
表3.6列出了序列TGGAAATAAAACGTAAGTAG 中所有碱基2字码(k=2)的初始位置和字码值。对于完全重复、长度大于2的同向重复或亚序列的搜索可只限于2字码重复的初始位置。在本例中只有4个重复的2碱基重复序列。例如,在位置4、5、8、9、10和15均发现了字码值为1的碱基重复序列。从有重复的第2个碱基为起点的3字码值及位置列于表3.7,其中发现字码值为1、45和49的序列有重复。以每一重复的3碱基为起点的4字码搜索未能发现更长的重复序列。
因此最长的同向重复为4、8、9位置上的AAA,13、17位置上的GTA以及7、14位置上的TAA。同样对图3.1鸡β球蛋白DNA序列进行同向重复序列搜索,一些最长同向重复序列列于表3.8。
表3.6 序列TGGAAATAAAACGTAAGTAAGTAG的2字码值和位置(Karlin,1983) 字码值碱基位置字码值碱基位置
1 4,5,8,9,10,15 9 3
2 11 10 -
3 16,19 11 2
4 6 12 13,17
5 - 13 7,14,18
6 - 14 -
7 12 15 1
8 - 16 1
表3.7 序列TGGAAATAAAACGTAAGTAG的3字码值和位置(Karlin, 1983) 字码值 碱基位置
1 4,8,9
2 10
3 15
4 5
45 13,17
49 7,14
51 18
表3.8 鸡β球蛋白DNA序列中(图3.1)长度为8或8以上的碱基重复序列 长度 重复序列 起始位置
8 GCCCTGGC 79,418
GCCAGGCT 85,377
CCAGGCTG 86,378
CAGGCTGC 87,379
TCCTTTGG 130,208
CCTTTGGG 131,209
TGGTCCGC 176,398
GGTCCGCG 177,399
9 GCCAGGCTG 85,377
CCAGGCTGC 86,378
TCCTTTGGG 130,208
TGGTCCGCG 176,398
10 GCCAGGCTGC 85,377
Karlin等(1983)提出了序列内存在的最长同向重复序列的统计显著性评价
方法。在核苷酸的位置为独立的假定下(相当于阶次为0的马尔科夫链),长度为n的序列中,最长同向重复Ln 的期望长度和方差为:
1)
/1ln()
1ln(ln 26359.0??++=
p p n L μ
2
2)
(ln 645
.1P L
=σ (3.1) 其中,P 为序列中碱基频率的平方和:
∑==4
12i i P P 用尽可能接近最大长度的期望均值的字码(即R→μL ) 来开始同向重复序列的搜索计算可能节省计算量。
可以用一个近似方法来验证以上统计假说。假定同向重复序列的长度呈正态分布。对于图3.1鸡β蛋白序列,A、C、G、T 四个碱基的次数分别为87、144、118和89,因而P=0.2614,最长重复序列的期望长度为8.13且具有期望方差0.9138。根据95%的正态分布概率,理论上可以预期最长同向重复序列不超过10。
四. DNA 序列的几何学分析—Z 曲线
DNA 序列实际上是一种用4种字母表达的“语言”,只是其“词法”和“语法”规则目前还没有搞清楚。人类的语言有文字、声音两种基本表现形式,此外还有手语、旗语甚至图画语等特殊表达形式。同样,DNA 序列作为一种语言,其表达形式也不是唯一的。传统上,DNA 序列是用4种字母符号表达的一维序列。这是一种抽象形式,适合于存储、印刷和代数算法的处理,包括比较、排列和查找特殊序列等。我国学者张春霆等开展了DNA 序列三维空间曲线表示形式,即DNA 序列几何表示形式的研究。几何形式虽然与符号形式完全等价,但显示了DNA 序列的新特征。两种形式各有其特点,相互补充。这一新方法,为解读DNA 序列信息提供了崭新的手段。
他们的研究始于对4种碱基对称性的观察,提出了用正面体表示碱基对称性。1994年,他们利用这种形式来表示任意长度的DNA 序列。现将这种序列表示方法简述如下。
考察一个长为L的单股DNA序列,方向(5'→3'或3'→5')不限。从第一个碱基开始,依次考察此序列,每次只考察一个碱基。当考察到第n个碱基时(n=1,2,…,L),数一下从1到n这个子序列中四种碱基各自出现的次数。设4种碱基A、C、G、T出现的次数分别以A n、C n、G n、T n 表示之,这里下标“n ”是表明这些整数是从1到n这个子序列中数出来的,如图3.2所示。显然,它们都是正整数。根据正四面体的对称性可以证明,在正面体内存在唯一的一个点P n 与这四个正整数对应。点P n 构成了四个正整数的一一对应映射。点P n 坐标可用四正整数表达:
x n =2(An+Gn)-n,
y n =2(An+Gn)-n, (3.2) z n =2(An+Tn)-n,
x n ,y n ,z n ∈[-n,n],n=1,2,…,L,
5’’
图 3.2 DNA序列示意图
其中x n,y n和z n为点P n的三个坐标分量。当n从1跑到L时,我们依次得到P1,
P
2,P
3
,…,P
L
共L个点。将相邻两点用适当的曲线连接所得到的整条曲线,就称
为表示DNA序列的Z曲线。可以证明,Z曲线与所表示的DNA序列是一一对应的,即给定一DNA序列,存在唯一的一条Z曲线与之对应;反之,给定一条Z曲线,可找到唯一的一个DNA序列与之对应。换言之,Z曲线包含了DNA序列的全部信息。Z 曲线是与符号DNA序列等价的另一种表示形式,一种几何形式。可以通过Z曲线对DNA序列进行研究。
Z曲线的三个分量(方程3.2)具有明确的生物学意义:x
n
表示嘌呤/嘧啶碱基沿序列的分布。当从1到n的这个子序列中(图3.2)嘌呤碱基多于嘧啶碱基时,
x
n >0,否则,x
n
<0,当两者相等时x
n
=0。同样,y
n
表示氨基/酮基碱基沿序列的分
布。当在子序列中氨基碱基多于酮基碱基时,y
n >0,否则,y
n
<0,当两者相等时
y
n =0。z
n
表示强/弱氢键碱基沿序列的分布。当弱氢键碱基多于强氢键碱基时,
z
n >0,否则z
n
<0,当两者相等时,z
n
=0。这三种分布是相互独立的,表现在以下
事实上:任何一种分布不能由其它两种分布的线性叠加表示出来。给定的DNA序列唯一地决定了这三种分布;三种分布唯一地描述了DNA序列。对DNA序列的研究就是通过对这三种分布的研究来进行。从方法学的角度来看,这是DNA序列的一种几何学研究途径。
图3.3给出了大肠杆菌ayoP基因族序列Z曲线的三个分量,即三种分布图。该基因族包含了大肠杆菌5 个基因aroP,A,aceFE,aceF和lpd,总长度为9501bp,分别编码芳香族氨基酸运输蛋白aroP,蛋白质A(功能不详)和三种酶,即丙酮酸脱氢酶,二氢硫辛酰基转移酶和二氢硫辛酰脱氢酶。它们位于此序列的0039-1406,1947-2654,2870-5527,5545-7434,7759-9183区间。在图中X轴的下方的基因排列图上已分别用阴影标出相应基因。在这些基因之间有三个启动子区(pm1、pm2和pm3),其中aceE和aceF基因属于ace操纵子,共用一个启动子。三个启动子区亦在图中标出。非常令人感兴趣的是,在5个编码区,Z曲线的z分量基本上都是单调下降的,而在三个启动子区基本上都是单调上升的。x,y分量亦有变化,但不如z分量明显。在上升、下降的交界处,Z曲线均发生了重大的转折,据此有可能用Z曲线识别这些位置。由此图可见,用Z曲线这种几何方法显示DNA序列不仅直观,而且作为一种识别序列中的不同基因和功能区的新方法,展现了广阔的应用前景。
图3.3 大肠杆菌ayoP基因族序列Z曲线的三个分量(三种分布图) 第二节 序列联配2
一.Needleman-Wunsch算法
有2种经典方法可以计算两条序列间的最适联配。Needleman-Wunsch算法是一种整体联配(global alignment)算法,最佳联配中包括了全部的最短匹配序列。Smith-Wateman算法是在Needleman-Wunsch算法基础上发展而来的,它是一种局部联配(Local alignment)算法。这二种算法均可以用于核酸和蛋白质序列。在给定空位罚值和替换矩阵情况下,它们总是能给出具有最高(优)联配值的联配。但是,这个联配并不需要达到生物学意义上的显著水平。GCG软件包中,BESFIT和GAP程序,EMBOSS的needle等可用于该联配。一些网站可以通过递交序列进行两条序列的联配分析。
从整体上分析两个序列的关系,即考虑序列总长的整体比较,用类似于使整体相似 (global similarity)最大化的方式,对序列进行联配。两个不等长度序列的联配分析必需考虑在一个序列中圈掉一些碱基或在另一序列作空位(gap)处理。Needleman 和Wunsch(1970)的法则为这些步骤提供了实例。这一算法是为氨基酸序列发展的,但也可以用于核苷酸序列。算法最初寻求的是使两条序列间
2部分内容取自Weir B.S.Genetic Data AnalysisⅡ—Methods for Discrete Population Genetic Data, Sunderland:Sinauer Associates Inc.Publishes,
1996
的距离最小。尽管这类距离的元素是以一种特定的方式定义的,但该算法的良好特性在于它确定了最短距离。这是一个动态规划(dynamic programming)的方法。
将两条联配的序列沿双向表的轴放置,两条序列的所有可能的联配方式都将在它们所形成的方形图中(见下图)。从任一碱基对,即表中的任一单元开始,联配可延三种可能的方式延伸:如果碱基不匹配,则每一序列加上一个碱基,并给其增加一个规定的距离权重;或在一个序列中增加一个碱基而在另一序列中增加一个空位或反之亦然。引入一个空位时也将增加一个规定的距离权重。因此,表中的一个单元可以从(至多)三个相邻的单元达到。我们把达左上角单元距离最小的方向看作相似序列延伸的方向。等距离时意味着存在两种可能的方向。将这些方向记录下来,并在研究了所有的单元之后,沿着记录的方向就有一条路径可从右下角(两个序列的末端)追踪到左上角 (两个序列的起点)。由此所产生的路径将给出具有最短距离的序列联配。
以两个短序列CTGTATC和CTATAATCCC为例,将上述过程说明于图3.4。设碱基错配时距离权重为1,引入一个空位时距离权重为3。该图边缘的行和列作为起始条件增加到表中。在单元5行3列,即相应较短序列(第二序列)的第2个T碱基和较长序列(第一序列)的第1个T碱基位置,有三种可能的距离增量。设在各序列中增加碱基T时 (从4行2列移动)对距离的贡献为0。从5行2列的位置作水平移动(等价于增加第二序列的碱基T而在第一序列引入一个空位),在本例中增加一个罚值3。从3列4行向该单元作垂直移动,使第一序列增加碱基T而第二序列引入一个空位,结果也得到一个罚值3。因此从该单元(5行3
列)所得到的最小距离的延伸方向是沿对角线和水平方向。在表中这两个方向用箭头表示。这两种最短方向都使从左上角到该单元的距离为6。沿箭头所指方向在表中从右下角向左上角追踪,得到6种可能的联配:
CTATAATCCC CTATAATCCC CTATAATCCC
CTGTA-TC-- CTGTA-T-C- CTGTA-T--C
CTATAATCCC CTATAATCCC CTATAATCCC
CTGT-ATC-- CTGT-AT-C- CTGT-AT--C
在上述6种联配中,距离均为10,即在较短序列中有6个匹配碱基、1个错配碱基和3个空位。
https://www.wendangku.net/doc/645065839.html,/bioinplant/ 《生物信息学札记》 樊龙江
0 C T A T A A T C C C 0 2 5 8← 1 4 7 0←33←36←39←31←31←3132←32←32←330 ↑ 2 5← 8 1 4 ↑ 2← 5 8 1 ↑ ← 0← 3 6 9 ↑ ↑ ← 0 3 6 ↑ ↑ 2 ← 0 3 ↑ ↑ ↑ 5 0 ↑ ↑ ↑ ↑ ↑ ↑ C 3 0 3 1 3 1 3 1 3 1 3 1 3 1 3 0 3 0 3 0 3 330←33←36←39←31←3131←32←32←327↑ T 3 1 3 0 3 1 3 0 3 1 3 1 3 0 3 1 3 1 3 1 3 63330←33←36←39←3131←31←32←324↑ ↑ G 3 1 3 1 3 1 3 1 3 1 3 1 3 1 3 1 3 1 3 1 3 9363331←34←37←3131←31←31←322↑ ↑ T 3 1 3 0 3 1 3 0 3 1 3 1 3 0 3 1 3 1 3 1 3 1239363431←34←3731←31←31←319↑ ↑ A 3 1 3 1 3 0 3 1 3 0 3 0 3 1 3 1 3 1 3 1 3 153139363431←3437←31←31←316↑ ↑ T 3 1 3 0 3 1 3 0 3 1 3 1 3 0 3 1 3 1 3 1 3 18313123936343234←37←31←313↑ ↑ C 3 0 3 1 3 1 3 1 3 1 3 1 3 1 3 0 3 0 3 0 3 21 3 18 3 15 3 12 3 9 3 7 3 5 3 3 3 4 ← 3 7 ← 3 10
图3.4 Needleman-Wusch 算法实例。设定碱基错配的距离权重为1,单个碱基缺失或插入时距离权重为3
34
该算法可以用代数形式来描述。设具有碱基a i 和b j 的两个序列a和b,这两个序列间距离为d(a,b)。通过评价序列a中前i个位置和序列b前j位置的距离
,递归地得到距离d(a,b)。如果a和b的长度为m和n,则其期望距离为。上表中引入的第1行1列单元的距离为0(相当于空序列),在单元(i,j)内,使到达该单元距离增加的三种可能事件为:
),(j i b a d ),(n m b a d 1.从单元(i-1,j)向(i,j)的垂直移动,相当于在b序列中插入一个空位使相似序列延伸。换言之,b序列由a序列中a i 的缺失所产生,这一事件的权重记作。 )_(i a w 2.从单元(i-1,j-1)向(i,j)的对角线移动,相当于增加碱基a i 和b j 使相似序列延
伸。换言之,b序列由a序列中的a i 被b j 取代所产生,这一事件的权重记为
。
),_(j i b a w 3.从单元(i,j-1)向(i,j)的水平移动,相当于在序列b中插入一个空位使相似序列延伸。换言之,b序列由b j 插入a序列所产生,这一事件的权重记为。
)(j b w +因此,单元(i,j)的距离可看成三个相邻单元的距离加上相应权重后的最小者,即
),(j i b a d (3.3)
??
?
??+++=+?????)(),(),(),()(),(min ),(1111i j i j i j i i j i j i b w b a d b a w b a d a w b a d b a d 且初始条件为
0),(00=b a d
∑∑=?=+==i
k k i j
k k j
a w
b a d b w b a d 1
010
)
(),()
(),(在图3.4的实例中
(对于每一个i)
3)(=?i a w ???=10
),(j i b a w )()(j i j i ≠=
(对于每一个j)
3)(=+j b w 当两个序列被联配时,通过计算其重排序列(shuffed version)的联配距离,
可以得到这两个序列间的最小距离估计。如果实际得到的联配距离小于重排序列距离的95%,则表明实际的联配距离达到了5%的显著水平,是不可能由机误造成的。
二.Smith-Waterman 算法
由于亲缘关系较远的蛋白质序列可能只有一些相互独立的相同片段,所以进行局部相似性分析有时可能比整体相似性分析更合理。Smith和Waterman描述了一种查找具有最高相似性片段的算法。对于序列A=(a 1,a 2,…,a m )和 B=(b 1,b 2,…,b n ),H ij 被定义为以a i 和b j 碱基对结束的片段(亚序列)的相似性值。与Needle-Wunsch算法一样,Smith-Waterman算法也要利用递推关系来确定H值,H的初始值为:
, 0≤i ≤n, 00=i H 00=j H , 0≤j ≤m
相似性计算中包括2个统计量:碱基对(序列因子) 的相似性值
和空位权重(k 为空位长度)。Smith-Waterman 算法可以给出2条序列的最大相似性值。以碱基对结束的片段可以由以和结束片段增加碱基(因子)来获得,或者可以删除k 长度的碱基片段,可删除l 长度碱基片段。具体算法如下:
j i b a ,),(j i b a S uk v w k +=j i b a ,1?i a 1?j b i a j b
)
,max(),max(1,11,,11,1u P w H Q u P w H P j i j i ij j i j i ij ??=??=???? (3.4)
则 ,(1≤i≤m,1≤j≤n) (3.5)
?????
???=?=+=?≤≤?≤≤??0)(max )
(max )
,(max ,1,11,1l l j i j l ij k j k i i k ij j i j i ij w H Q w H P b a S H H 其中 00,0,0,00,0====i j Q Q P P
该算法可以确保具有最大H ij 值的序列片段是相似性最好的。从为起点,向后追踪矩阵,直到到达某一负值。对于具有最大相似性片段以外部分的差异性不会影响到该片段的H值。
),(j i b a ij H 举例说明了这一算法。我们同样以上节Needleman-Wunsch 算法中的两条短序列为例。两条序列(CTGTATC 和CTATAATCCC)排于表3.9的两侧,相应的,和值分别列入表中。本例的权重等根据Smith 和Waterman(1981)以前的例子设定为:
ij H ij P ij Q
????=3
/11
),(j i b a S j
i j i b a b a ≠=
=1+k/3 (3.6) k w 对于4个碱基具有相同频率的随机长序列,值的平均值为零。值应至少不小于匹配与不匹配权重的差值。
),(j i b a S k w 表3.9的最大H ij 为4.33(8行与7列相交处),星号(*)表示出具有最大相似性的片段匹配方式:
CTGTA-TC CTATAATC
表3.9 Smith-Waterman算法例举
j=0 j=1j=2j=3j=4j=5 j=6 j=7
0 C T G T A T G i=0 0
H0 00000 0 0 ij
P0 00000 0 0
ij
Q0 00000 0 0 ij
i=1 C
H0 1.00*0.000.000.000.00 0.00 1.00 ij
P0 -0.33-0.33-0.33-0.33-0.33 -0.33 -0.33
ij
Q0 -0.33-0.33-0.67-1.00-1.33 -1.33 -1.33 ij
i=2 T
H0 0.00 2.00*0.67 1.000.00 1.00 0.00 ij
P0 -0.33-0.67-0.67-0.67-0.67 -0.67 -0.33
ij
Q0 -0.33-0.670.670.330.00 -0.33 -0.33 ij
i=3 A
H0 0.000.67 1.67*0.33 2.00 0.67 0.67 ij
P0 -0.670.67-0.67-0.33-1.00 -0.33 -0.67
ij
Q0 -0.33-0.67-0.670.330.00 0.67 0.33 ij
i=4 T
H0 0.00 1.000.33 2.67* 1.33 3.00 1.67 ij
P0 -1.000.330.33-0.670.67 -0.67 -0.67
ij
Q0 -0.33-0.67-0.33-0.67 1.33 1.00 1.67 ij
i=5 A
H0 0.000.000.67 1.33 3.67* 2.33 2.67 ij
P0 -1.330.000.00 1.330.00 1.67 0.33
ij
Q0 -0.33-0.67-1.00-0.670.00 2.33 2.00 ij
i=6 A
H0 0.000.000.00 1.00 2.33* 3.33 2.00 ij
P0 -1.33-0.33-0.33 1.00 2.33 1.33 1.33
ij
Q0 -0.33-0.67-1.00-1.33-0.33 1.00 2.00 ij
i=7 T
H0 0.00 1.000.00 1.00 2.00 3.33* 3.00 ij
P0 -1.33-0.67-0.670.67 2.00 2.00 1.00
ij
Q0 -0.33-0.67-0.33-0.67-0.33 0.67 2.00 ij
i=8 C
H0 1.000.000.670.33 1.67 2.00 4.33* ij
P0 1.33-0.33-1.000.33 1.67 2.00 1.67
ij
Q0 -0.33-0.33-0.67-0.67 1.00 0.33 0.67 ij
i=9 C
H0 1.000.670.000.33 1.33 1.67 3.00 ij
P0 -0.33-0.67-0.670.00 1.33 1.67 3.00
ij
Q0 -0.33-0.33-0.67-1.00-1.00 0.00 0.33 ij
i=10 C
H0 1.000.670.330.00 1.00 1.33 2.67 ij
P0 -0.33-0.67-1.00-0.33 1.00 1.33 2.67
ij
Q0 -0.33-0.33-0.67-1.00-1.33 -0.33 0.00 ij
三.序列相似性的统计特性
3
到目前为止,对局部联配的统计学问题已基本搞清楚,特别是那些不含有空位(gap)的局部联配更是如此。我们不妨首先考虑不含有空位的局部联配问题,BLAST 最初的搜索程序便是以此为基础的。
无空位局部联配涉及的是等长度的一对序列片段,两个片段的各部分彼此比较。一种Smith-Waterman 或Sellers 算法的改进算法可以找到所有高比值片段对(high-scoring segment pairs,HSPs),即这些片段对的比较分值不会因片段的延伸而进一步升高。
为了分析上述分值随机性产生的几率大小,需要建立一个随机序列模型。对于蛋白质而言,最简单的序列模型可通过从一条序列中随机地选取氨基酸残基,当然这一条序列中各种残基的频率必需一定。另外,一对随机氨基酸的联配期望值必需为负值,否则不论联配片段是否相关的,都会得到高比值,统计理论也将派不上用场。
就象独立随机变量之和总是倾向于正态分布(normal distribution)一样,独立随机变量的最大值倾向于极值分布(extreme value distribution)。在研究最佳局部联配时,主要涉及的是后一种情况。在一定的序列长度m 和n 限定下,HSP 的统计值可由2个参数(k 和λ)确定。最简单的形式,即不小于比较值为S 的HSP 个数,可由下列公式算得其期望值:
(3.7)
s kmne E λ?=我们称该期望值为比值S 的E 值(E-Value)。
上述公式非常灵敏。在给定比值的情况下,将比较序列长度加倍,则HSP 数(即E 值)也将加倍,同样,S 值为2X 的某个HSP 长度必是S 值为X 的两倍,所以E 值将随着s 值的增大急剧减少。参数K 和λ可分别被简单地视为搜索步长(search spacesize)和计分系统(scoring system)的特征数。
1.二进制值或标准比值(Bit score)
最初获得的比值(S)在没有计分系统或统计量K 和λ的辅助下,没有什么意义。单独的比值就如同没有单位(米或者光年)的距离。可使比值按下式标准化:
2
ln ln k
s S ?=
′λ (3.8) 获得S ’
值就如同得到了具有标准单位的数值。 E 值因此可简化为:
(3.9) S mn E ′?=2二进制值使所使用的计分系统赋予了统计学意义,使除了可以确定搜索步长
外,同样可以计算相应的显著水平。 2.P 值(P-Value)(概率值)
具有大于或等于某一比值S 的随机HSP 数可由泊松分布(Poisson
distribution)确定。由此可以计算出搜索到某一比值大于或等于S 的HSP 的机率为
3
译自NCBI BLAST TURORIAL:The statistics of sequence similarity scores.
!
X E e
X
E
? (3.10) 式中E 由(3.7)式确定。
作为一个特例,搜索不到比值≥S的HSP概率为e -E,
,
所以至少发现一个HSP (比值≥S)的概率为
(3.11)
)exp(11x E kmne e P λ????=?=这是与比值S 相关的P 值(概率值)。例如,在可能搜索到3个比值≥S 的HSP 的情况下,至少发现一个HSP 的机率为0.95[可由(3.11)式算得]。BLAST 程序中使用了E 值而非P 值,这主要是从直观和便于理解的角度考虑。比如E 值等于5和10,总比P 值等于0.993和0.99995更直观。但是当E<0.01时,P 值与E 值接近相同。
3.数据库搜索策略
E 值计算公式[公式(3.7)]可以应用于2个蛋白质序列长度分别为m 和n 的比较,但是对于某一序列长度为m 的蛋白序列,如何在那些长短不一的数据库序列中找到与之匹配良好的序列呢?一种思路是把数据库中的所有蛋白序列与待查序列的关系都视为相同重要,也就是说对于E 值均较低的短和长序列,它们是等同重要的。FASTA 程序近期版本便是采用这一策略。另一种思路是把长序列视为比短序列更重要,因为长序列往往包括更多的特异功能域(domain)。如果对序列长度上进行相关优先处理,则在计算数据库序列长度为n 的E 值时,将乘以N/n,其中N 为数据库中序列的总长度。根据公式(3.7),E 值的计算可简单地把整个数据库序列视为长度为N 的单条序列。BLAST 程序采用了这一策略。FASTA 策略中E 值的计算还需再乘上数据库的序列条数。如果考虑到核酸数据库的序列长度变化更大,则在DNA 序列相似性搜索时,BLAST 的策略可能会是合理的选择。
一些数据库搜索程序,例如FASTA 或其它基于Smith-Waterman 算法的程序,在进行序列搜索时,会对数据库中的每条序列进行联配并给出联配值,这些值大部分与未知序列无关,但它们被用于了K 和λ参数的估计。这一方法避免了随机序列模型因使用真实序列(real sequence)造成的随意性,但同时产生了使用相关序列估计参数的难题。BLAST 仅通过部分而不是全部无关序列计算最适联配值,这赢得了搜索速度。因此,对于某一选定的替换矩阵和空位罚值,必须进行K 和λ参数的预先估计,估计中使用真实序列,而非通过随机序列模型产生的模拟序列。这一估计的结果看来非常准确。 4.空位联配(gapped alignment)的统计问题
根据统计理论,以上述及的统计方法只适用于不含有空位的局部联配(非空位联配)。但是,许多计算试验和分析结果充分证明,上述统计方法同样适用于空位联配。对于非空位联配,可用基于替换矩阵和比较序列的残基频率的办法估计统计参数;对于空位联配,参数的估计则必须根据“随机”序列的大尺度比较。 5.边际效应(edge effect)
以上统计学方法对于短序列来说有些偏差。这些统计方法的基础理论是一个渐近理论,该理论假设局部联配可以适用于任何规模的联配。但是,一个高比值联配必须有一定的长度,不能从接近二条序列末端的地方开始。这种边际效应可以通过计算序列的“效应长度”(effective length)来修正。BLAST 程序中包含了这一修正过程。对于长于200残基的序列可以不进行边际效应的修正。
6.替换矩阵的选择
局部联配的结果与所选用的替换矩阵紧密相关。没有任何一个计分方案(即替换矩阵)可以适用于所有研究目标,对于局部联配的计分基础理论的正确理解可以极大促进序列分析准确性。相关内容详见第4小节。 7. 空位罚值(gap penalties)
联配中另一个重要问题是空位问题。空位处理是针对序列进化过程中可能发生的插入和缺失而设计的。插入和缺失可能只涉及1个或2个残基,也可能是整个功能域(domain),所以,在进行空位罚值设计时必须反映这些情况。
有2个参数应用于空位罚值设定,一个与空位设置(gap opening)有关,另一个与空位扩展(gap extension)有关。任一空位的出现均处以空位设置罚值,而任一空位的扩大必须处于空位扩展罚值。对于一个空位长度为k的罚值W K 可用下式表示:
(3.12)
bk a w k +=其中a 是空位设置罚值,b 为空位扩展罚值。这两个参数值设置的变化对联配产生影响(表3.10).
表3.10 空位设置和空位扩展罚值对联配的影响
空位设置罚值 (Gap opening penalty) 空位扩展罚值 (Gap extension
penalty)
说 明
(Comment)
大 大
极少插入或缺失:适用于非常相关蛋白质间的联配;
大
小
少量大块插入:用于整个功能域可能插入的情况
小
大
大量小块插入:适用于亲缘关系较远的蛋白质同源性分析
经过多年的试验,一个合适的空位罚值已经被确定下来。大多数联配程序均对特定的替换矩阵设定了空位罚值的缺略值(default),如果使用者希望使用不同的替换矩阵,则原来的空位罚值设定不一定合适。如何设定罚值并无明确的理论可遁,但大的空位设置罚值配以很小的空位扩展罚值被普遍证实是最佳的设定思路。
四. 替换矩阵
4
1.替换矩阵的一般原理
我们并不能直接计算出两条序列的最佳联配,我们需要找到一个可以估计任何联配的某一统计数,使生物学关系匹配最显著的联配统计数最大。
4
本部分内容主要取自Weir B.S.(徐云碧等译). 遗传学数据分析—群体遗传学离散型数据分析方法,北京:中国农业出版社,1996
先看以下2条氨基酸序列的联配情况。如果我们将各残基按相同的统计数处理,则2种联配(a和b)的得分将是相等的(9个残基中5个匹配):
(a) TTYGAPPWCS (b) TTYGAPPWCS
TGYAPPPWS TGYAPPPWS
* *** * * * ***
但是联配a是一些相对普通的残基(A、P、S和T)保持一致,而联配b则是一些相对稀有残基(W-色氨酸、Y-酪氨酸)相一致。我们需要一个更科学的赋分方法来反映匹配氨基酸间生物学和化学关系。
在联配中,C-C匹配相对比S-S匹配更重要些,因为半胱氨酸(C)是具有非常特殊性质的相对稀有氨基酸,而丝氨酸(S)则相对普通。同样D-E匹配应取正值,因为这两个残基具有相同的化学性质,在两条联配的蛋白质序列中能起到相同的功用。但是,V-K匹配则应被罚分,因为这两个残基毫无相似,不可能在两条序列中引到一样的作用。
替换矩阵(substitution matrices)包括了在联配中各种匹配方式如何赋分的信息,故替换矩阵又常被称为计分矩阵(scoring matrices)。
用于DNA序列联配的替换矩阵相对比较直观。以下是一个常被使用的替换矩阵:
A C G T
A 0.9 -0.1 -0.1 -0.1
C -0.1 0.9 -0.1 -0.1
G -0.1 -0.1 0.9 -0.1
T -0.1 -0.1 -0.1 0.9
矩阵中每个匹配的碱基对均计为0.9分,每个不匹配的碱基对被罚0.1分,这样,下面一个联配的得分应为4.3(=5×0.9+2×(-0.1)):
GCGCCTC
GCGGGTC
*** **
用于蛋白质联配的替换矩阵要复杂一些,因为没有一个矩阵可以适用各种情况。构建矩阵时应考虑不同的蛋白质家族在进化过程中,一种氨基酸突变成另一种氨基酸概率的差异,根据不同的蛋白质家族和预期的相似程度构建不同的替换矩阵。2个最有名的蛋白质替换矩阵是PAM和BLOSUM,它们分别是在1979年和1992年完成的。
最后,一个重要的概念必须明确。同源性(homology)和相似性(similarity)是不同的2个概念,不能混淆和混用。2条序列具有同源性,意味着这两条序列有进化方面的关系,它们从一条共同的祖先序列进化而来;而相似性,只是表明一种相似程度。
2.PAM氨基酸替换矩阵
在进行蛋白质序列联配时,必须通过一定的方法给联配的残基对赋予一定的分值,替换矩阵便是其中最重要的方法。
已故Dayhoff是蛋白质列序比较的先驱,她和她的同事们通过对蛋白质进化模式的研究,建立了一组被广泛应用的替换矩阵,这些矩阵常被称为Dayhoff,MDM(Mutation Data Matrix)或PAM(Percent Accepted Mutation)矩阵。
应用于DNA序列的许多算法最初是从氨基酸蛋白质序列的一些算法发展而来的。由于蛋白质最有可能是自然选择的目标,可以认为蛋白质序列的分析比DNA 分析更具有生物学意义。蛋白质分析完全避免了几个三联体可能编码同一氨基酸的遗传密码简并问题。有必要进一步分析各种氨基酸间的同源性程度,以及在进化过程中一种氨基酸被另一种氨基酸替换的概率大小。也许把氨基酸按一定特性分成若干组更便于以上分析,例如氨基酸可分成中性疏水(G、A、V、L、I、F、P、M)、中性亲水(S、T、Y、W、N、E、C)、碱性(K、R、H)和酸性(D、E)氨基酸等。在比较许多具有相似性蛋白质序列的基础上,Dayhoff等于1979年构建了一个突变概率矩阵M(mutation probability matrix)。最初她们比较了许多对蛋白质序列,以确定进化过程中一种氨基酸被另一种氨基酸取代的经验资料。她们共观测到1572次取代“事件”。以此为基础,她们建立了表3.11的“可观测点突变矩阵”A(accepted point mutation matrix)(由于舍入误差使表中的数值相加不完全等于1572)。氨基酸i被氨基酸j替换的经验次数(记作A ij )可从上表中找到。矩阵A可被称为原始PAM矩阵。
由矩阵A可以进一步获得突变概率矩阵M。矩阵M的元素M ij 表示经过一定的进化时期氨基酸j被氨基酸i所替换的经验频率。Dayhoff等进而把可观测突变百分率(percent accepted mutation或point accepted mutation per 100 residues),即PAM作为一种时间度量单位。假设同一位点不会发生二次以上的突变,则1PAM 等于100个氨基酸多肽链中预期发生一次替换所需的时间。
Dayhoff 提出了一个称为相对“突变力”(mutability)的概念,并将氨基酸j 的相对突变力定义为观测到的氨基酸突变数除以联配序列中j 氨基酸的频率,即:
(3.13)
∑≠∝j
i j ij j f A m /这里将氨基酸a j 所有可能的变化均考虑在内。各种氨基酸的m j 和f j 值(经标准化)列于表3.12。
生物信息学软件及使用概述
生物信息学软件及使 刘吉平 liujiping@https://www.wendangku.net/doc/645065839.html, 用概述 生 物秀-专心做生物! w w w .b b i o o .c o m
生物信息学是一门新兴的交叉学生物信息学的概念: 科,它将数学和计算机知识应用于生物学,以获取、加工、存储、分类、检索与分析生物大分子的信息,从而理解这些信息的生物学意义。 生 物秀-专心做生物! w w w .b b i o o .c o m
分析和处理实验数据和公共数据,生物信息学软件主要功能 1.2.提示、指导、替代实验操作,利用对实验数据的分析所得的结论设计下一阶段的实验 3.实验数据的自动化管理 4.寻找、预测新基因及其结构、功能 5.蛋白质高级结构及功能预测(三维建模,目前研究的焦点和难点) 生 物秀-专心做生物! w w w .b b i o o .c o m
功能1. 分析和处理实验数据和公共数据,加快研究进度,缩短科研时间 ?核酸:序列同源性比较,分子进化树构建,结构信息分析,包括基元(Motif)、酶切点、重复片断、碱基组成和分布、开放阅读框(ORF ),蛋白编码区(CDS )及外显子预测、RNA 二级结构预测、DNA 片段的拼接; ?蛋白:序列同源性比较,结构信息分析(包括Motif ,限制酶切点,内部重复序列的查找,氨基酸残基组成及其亲水性及疏水性分析),等电点及二级结构预测等等; ?本地序列与公共序列的联接,成果扩大。 生 物秀-专心做生物! w w w .b b i o o .c o m
Antheprot 5.0 Dot Plot 点阵图 Dot plot 点阵图能够揭示多个局部相似性的复杂关系 生 物秀-专心做生物! w w w .b b i o o .c o m
生物信息学题库
■一、选择题: 1.以下哪一个是mRNA条目序列号: A. J01536■. NM_15392 C. NP_52280 D. AAB134506 2.确定某个基因在哪些组织中表达的最直接获取相关信息方式是:■. Unigene B. Entrez C. LocusLink D. PCR 3.一个基因可能对应两个Unigene簇吗?■可能 B. 不可能 4.下面哪种数据库源于mRNA信息:■dbEST B. PDB C. OMIM D. HTGS 5.下面哪个数据库面向人类疾病构建: A. EST B. PDB ■. OMIM D. HTGS 6.Refseq和GenBank有什么区别: A. Refseq包括了全世界各个实验室和测序项目提交的DNA序列B. GenBank提供的是非冗余序列 ■. Refseq源于GenBank,提供非冗余序列信息D. GenBank源于Refseq 7.如果你需要查询文献信息,下列哪个数据库是你最佳选择: A. OMIM B. Entrez ■PubMed D. PROSITE 8.比较从Entrez和ExPASy中提取有关蛋白质序列信息的方法,下列哪种说法正确:A. 因为GenBank的数据比EMBL更多,Entrez给出的搜索结果将更多B. 搜索结果很可能 一样,因为GenBank和EMBL的序列数据实际一样■搜索结果应该相当,但是ExPASy中的SwissProt记录的输出格式不同 9.天冬酰胺、色氨酸和酪氨酸的单字母代码分别对应于:■N/W/Y B. Q/W/Y C. F/W/Y D. Q/N/W 10.直系同源定义为:■不同物种中具有共同祖先的同源序列B. 具有较小的氨基酸一致性但是有较大的结构相似性的同源序列 C. 同一物种中由基因复制产生的同源序列 D. 同一物种中具有相似的并且通常是冗余的功能的同源序列 11.下列那个氨基酸最不容易突变: A. 丙氨酸B. 谷氨酰胺 C. 甲硫氨酸■半胱氨酸 12.PAM250矩阵定义的进化距离为两同源序列在给定的时间有多少百分比的氨基酸发生改变: A. 1% B. 20%■. 80% D. 250% 13.下列哪个句子最好的描述了两个序列全局比对和局部比对的不同:A. 全局比对通常用于比对DNA序列,而局部比对通常用于比对蛋白质序列B. 全局比对允许间隙,而局 部比对不允许C. 全局比对寻找全局最大化,而局部比对寻找局部最大化■全局比对比对整体序列,而局部比对寻找最佳匹配子序列 14.假设你有两条远源相关蛋白质序列。为了比较它们,最好使用下列哪个BLOSUM和PAM矩阵:■BLOSUM45和PAM250 B. BLOSUM45和PAM 1 C. BLOSUM80和PAM250 D. BLOSUM10和PAM1 15.与PAM打分矩阵比较,BLOSUM打分矩阵的最大区别是:A. 最好用于比对相关性高的蛋白B. 它是基于近相关蛋白的全局多序列比对 ■它是基于远相关蛋白的局部多序列比对D. 它结合了全局比对和局部比对 16.如果有一段DNA序列,它可能编码多少种蛋白质序列: A. 1 B. 2 C. 3 ■. 6 17.要在数据库查询一段与某DNA序列编码蛋白质最相似的序列,应选择: A. blastn B. blastp C. tblastn D. tblastp■blastx 18.为什么ClustalW(一个采用了Feng-Doolittle渐进比对算法的程序)不报告E值:A. ClustalW报告E值■使用了全局比对 C. 使用了局部比对 D. 因为是多序列比对 19.Feng-Doolittle方法提出“一旦是空隙,永远是空隙”规则的依据是:A. 保证空隙不会引物序列加入而填充B. 假定进化早期分歧的序列有较高优先级别■假定最近序列空隙应 该保留 D. 假定最远序列空隙应该保留 20.根据分子钟假说:A. 所有蛋白质都保持一个相同的恒定进化速率 B. 所有蛋白质的进化速率都与化石记录相符合C. 对于每一个给定的蛋白质,分子进化的速率是逐 渐减慢的,就如同不准时的钟■对于每一个给定的蛋白质,其分子进化的速率在所有的进化分支上大致是恒定 21.系统发生树的两个特征是: A. 进化分支和进化节点■树的拓扑结构和分支长度C. 进化分支和树根D. 序列比对和引导检测方法 22.下列哪一个是基于字母特征的系统发生分析的算法:A. 邻位连接法(NJ法)B. Kimura算法■最大似然法(ML)D. 非加权平均法(UPGMA) 23.基于字母特征和基于距离的系统发生分析的算法的基本差异是:■基于字母特征的算法没有定义分支序列的中间数据矩阵 B. 基于字母特征的算法可应用于DNA或者蛋白质序列,而基于距离仅能用于DNA C. 基于字母特征的算法无法运用简约算法 D. 基于字母特征的算法的进化分支与进化时间无关 24.一个操作分类单元(OTU)可指:A. 多序列比对■蛋白质序列C. 进化分支D. 进化节点 25.构建进化树最直接的错误来源是:■多序列比对错误B. 采样的算法差异C. 假设进化分支是单一起源D. 尝试推测基因的进化关系 26.第一个被完整测定的基因组序列是:A. 啤酒酵母的3号染色体B. 流感病毒■ФX174 D. 人类基因组 27.普通的真核生物线粒体基因组编码大约多少个蛋白质:■10 B. 100 C. 1000 D. 10000 28.根据基因组序列预测蛋白质编码基因的算法的最大问题是:A. 软件太难使用■. 假阳性率太高,许多不是外显子的序列部分被错误指定C. 假阳性率太高,许 多不是外显子功能未知 D. 假阴性率太高,丢失太多外显子位点 29.HIV病毒亚型的系统演化研究可以:A. 证实HIV病毒是由牛病毒演化而来■. 用于指导开发针对保守蛋白的疫苗C. 证实哪些人类组织最容易遭受病毒侵染 30.一个典型的细菌基因组大小约为多少bp:A. 20000■. 200000 C. 2000000 D. 20000000
最新生物信息学考试复习
——古A.名词解释 1. 生物信息学:广义是指从事对基因组研究相关的生物信息的获取,加工,储存,分配,分析和解释。狭义是指综合应用信息科学,数学理论,方法和技术,管理、分析和利用生物分子数据的科学。 2. 基因芯片:将大量已知或未知序列的DNA片段点在固相载体上,通过物理吸附达到固定化(cDNA芯片),也可以在固相表面直接化学合成,得到寡聚核苷酸芯片。再将待研究的样品与芯片杂交,经过计算机扫描和数据处理,进行定性定量的分析。可以反映大量基因在不同组织或同一组织不同发育时期或不同生理条件下的表达调控情况。 3. NCBI:National Center for Biotechnology Information.是隶属于美国国立医学图书馆(NLM)的综合性数据库,提供生物信息学方面的研究和服务。 4. EMBL:European Molecular Biology Laboratory.EBI为其一部分,是综合性数据库,提供生物信息学方面的研究和服务。 5. 简并引物:PCR引物的某一碱基位置有多种可能的多种引物的混合体。 6. 序列比对:为确定两个或多个序列之间的相似性以至于同源性,而将它们按照一定的规律排列。
7. BLAST:Basic Local Alignment Search Tool.是通过比对(alignment)在数据库中寻找和查询序列(query)相似度很高的序列的工具。 8. ORF:Open Reading Frame.由起始密码子开始,到终止密码子结束可以翻译成蛋白质的核酸序列,一个未知的基因,理论上具有6个ORF。 9. 启动子:是RNA聚合酶识别、结合并开始转录所必须的一段DNA序列。原核生物启动子由上游调控元件和核心启动子组成,核心启动子包括-35区(Sextama box)TTGACA,-10区(Pribnow Box)TATAAT,以及+1区。真核生物启动子包括远上游序列和启动子基本元件构成,启动子基本元件包括启动子上游元件(GC岛,CAAT盒),核心启动子(TATA Box,+1区帽子位点)组成。 10. motif:模体,基序,是序列中局部的保守区域,或者是一组序列中共有的一小段序列模式。 11. 分子进化树:通过比较生物大分子序列的差异的数值重建的进化树。 12. 相似性:序列比对过程中用来描述检测序列和目标序列之间相似DNA碱基或氨基酸残基序列所占的比例。 13. 同源性:两个基因或蛋白质序列具有共同祖先的结论。
生物信息学课程设计
生物信息学课程设计报告 题目:用blast、clustalx2和mega来分析鼠伤寒沙门氏菌的四环素抗性基因 专业:生物技术 班级:11-2 学号:11114040235 姓名:邹炜球 指导教师:马超 广东石油化工学院生物工程系 2013年 12 月 21 日
摘要 生物信息学(Bioinformatics)是研究生物信息的采集,处理,存储,传播,分析和解释等各方面的一门学科,它通过综合利用生物学,计算机科学和信息技术而揭示大量而复杂的生物数据所赋有的生物学奥秘。本课程设计主要通过分析鼠伤寒沙门氏菌的四环素抗性基因来介绍生物信息学里面常用的数据库NCBI和一些常用的软件(如blast、clustalx2、Primer Premier 5和mega),由于生物信息学这一门课在生物研究领域所起到的作用非常大,所以熟练一些常用的生物信息学软件和数据库是非常有必要的。 关键词:NCBI、blast、clustalx2、Primer Premier 、mega、生物信息学、序列比对、系统发育树
目录 1绪论 (4) 1.1生物信息学的发展概况 (4) 1.2生物信息学的发展展望 (4) 2 课题设计内容 (5) 2.1以某一基因或蛋白为研究对象搜索一条序列(DNA长度为300-1500bp,蛋白质序列 为100-500)及相关信息,并分别表示出他的GENBANK和FASTA格式 (6) 2.2以设计内容1为目标序列进行BLAST分析 (7) 2.3通过BLAST或相关软件下载8条基因或蛋白质序列 (9) 2.4以8条基因序列进行多序列比对 (10) 2.5依照设计内容4构建系统发育树 (10) 2.6以其中一条基因序列设计一条长度为200-500bp的一对引物 (12) 参考文献 (16)
生物信息学名词解释资料
1.生物信息学:研究大量生物数据复杂关系的学科,其特征是多学科交叉,以互联网为媒介,数据库为载体。利用数学知识建立各种数学模型; 利用计算机为工具对实验所得大量生物学数据进行储存、检索、处理及分析,并以生物学知识对结果进行解释。 2.二级数据库:在一级数据库、实验数据和理论分析的基础上针对特定目标衍生而来,是对生物学知识和信息的进一步的整理。 3.FASTA序列格式:是将DNA或者蛋白质序列表示为一个带有一些标记的核苷酸或者氨基酸字符串,大于号(>)表示一个新文件的开始,其他无特殊要求。 4.genbank序列格式:是GenBank 数据库的基本信息单位,是最为广泛的生物信息学序列格式之一。该文件格式按域划分为4个部分:第一部分包含整个记录的信息(描述符);第二部分包含注释;第三部分是引文区,提供了这个记录的科学依据;第四部分是核苷酸序列本身,以“//”结尾。 5.Entrez检索系统:是NCBI开发的核心检索系统,集成了NCBI 的各种数据库,具有链接的数据库多,使用方便,能够进行交叉索引等特点。 6.BLAST:基本局部比对搜索工具,用于相似性搜索的工具,对需要进行检索的序列与数据库中的每个序列做相似性比较。P94 7.查询序列(query sequence):也称被检索序列,用来在数据库中检索并进行相似性比较的序列。P98 8.打分矩阵(scoring matrix):在相似性检索中对序列两两比对的质量评估方法。包括基于理论(如考虑核酸和氨基酸之间的类似性)和实际进化距离(如PAM)两类方法。P29 9.空位(gap):在序列比对时,由于序列长度不同,需要插入一个或几个位点以取得最佳比对结果,这样在其中一序列上产生中断现象,这些中断的位点称为空位。P29 10.空位罚分:空位罚分是为了补偿插入和缺失对序列相似性的影 响,序列中的空位的引入不代表真正的进化事件,所以要对其进行 罚分,空位罚分的多少直接影响对比的结果。P37 11.E值:衡量序列之间相似性是否显著的期望值。E值大小说明了 可以找到与查询序列(query)相匹配的随机或无关序列的概率,E 值越接近零,越不可能找到其他匹配序列,E值越小意味着序列的 相似性偶然发生的机会越小,也即相似性越能反映真实的生物学意 义。P95 12.低复杂度区域:BLAST搜索的过滤选项。指序列中包含的重复 度高的区域,如poly(A)。 13.点矩阵(dot matrix):构建一个二维矩阵,其X轴是一条序列, Y轴是另一个序列,然后在2个序列相同碱基的对应位置(x,y) 加点,如果两条序列完全相同则会形成一条主对角线,如果两条序 列相似则会出现一条或者几条直线;如果完全没有相似性则不能连 成直线。 14.多序列比对:通过序列的相似性检索得到许多相似性序列,将这 些序列做一个总体的比对,以观察它们在结构上的异同,来回答大 量的生物学问题。 15.分子钟:认为分子进化速率是恒定的或者几乎恒定的假说,从而 可以通过分子进化推断出物种起源的时间。 16.系统发育分析:通过一组相关的基因或者蛋白质的多序列比对或 其他性状,可以研究推断不同物种或基因之间的进化关系。 17.进化树的二歧分叉结构:指在进化树上任何一个分支节点,一个 父分支都只能被分成两个子分支。 系统发育图:用枝长表示进化时间的系统树称为系统发育图,是 引入时间概念的支序图。 18.直系同源:指由于物种形成事件来自一个共同祖先的不同物种中 的同源序列,具有相似或不同的功能。(书:在缺乏任何基因复制 证据的情况下,具有共同祖先和相同功能的同源基因。) 19.旁系(并系)同源:指同一个物种中具有共同祖先,通过基因重 复产生的一组基因,这些基因在功能上可能发生了改变。(书:由于 基因重复事件产生的相似序列。) 20.外类群:是进化树中处于一组被分析物种之外的,具有相近亲缘 关系的物种。 21.有根树:能够确定所有分析物种的共同祖先的进化树。 22.除权配对算法(UPGMA):最初,每个序列归为一类,然后找 到距离最近的两类将其归为一类,定义为一个节点,重复这个过程, 直到所有的聚类被加入,最终产生树根。 23.邻接法(neighbor-joining method):是一种不仅仅计算两两比 对距离,还对整个树的长度进行最小化,从而对树的拓扑结构进行 限制,能够克服UPGMA算法要求进化速率保持恒定的缺陷。 24.最大简约法(MP):在一系列能够解释序列差异的的进化树中 找到具有最少核酸或氨基酸替换的进化树。 25.最大似然法(ML):它对每个可能的进化位点分配一个概率, 然后综合所有位点,找到概率最大的进化树。最大似然法允许采用 不同的进化模型对变异进行分析评估,并在此基础上构建系统发育 树。 26.一致树(consensus tree):在同一算法中产生多个最优树,合并 这些最优树得到的树即一致树。 27.自举法检验(Bootstrap):放回式抽样统计法。通过对数据集多 次重复取样,构建多个进化树,用来检查给定树的分枝可信度。 精品文档
最新生物信息学复习题及答案(陶士珩)
生物信息学复习题 一、名词解释 生物信息学, 二级数据库, FASTA序列格式, genbank序列格式, Entrez,BLAST,查询序列(query),打分矩阵(scoring matrix),空位(gap),空位罚分,E 值, 低复杂度区域,点矩阵(dot matrix),多序列比对,分子钟,系统发育(phylogeny),进化树的二歧分叉结构,直系同源,旁系同源,外类群,有根树,除权配对算法(UPGMA),邻接法构树,最大简约法构树,最大似然法构树,一致 树(consensus tree),bootstrap,开放阅读框(ORF),密码子偏性(codon bias),基因预测的从头分析法,结构域(domain),超家族,模体(motif),序列表谱(profile),PAM矩阵,BLOSUM,PSI-BLAST,RefSeq,PDB数据库,GenPept, 折叠子,TrEMBL,MMDB,SCOP,PROSITE,Gene Ontology Consortium,表谱(profile)。 二、问答题 1)生物信息学与计算生物学有什么区别与联系? 2)试述生物信息学研究的基本方法。 3)试述生物学与生物信息学的相互关系。 4)美国国家生物技术信息中心(NCBI)的主要工作是什么?请列举3个以上NCBI 维护的数据库。 5)序列的相似性与同源性有什么区别与联系? 6)BLAST套件的blastn、blastp、blastx、tblastn和tblastx子工具的用途 什么? 7)简述BLAST搜索的算法。 8)什么是物种的标记序列? 9)什么是多序列比对过程的三个步骤? 10)简述构建进化树的步骤。 11)简述除权配对法(UPGMA)的算法思想。 12)简述邻接法(NJ)的算法思想。 13)简述最大简约法(MP)的算法思想。 14)简述最大似然法(ML)的算法思想。 15)UPGMA构树法不精确的原因是什么? 16)在MEGA2软件中,提供了多种碱基替换距离模型,试列举其中2种,解释其 含义。 17)试述DNA序列分析的流程及代表性分析工具。 18)如何用BLAST发现新基因? 19)试述SCOP蛋白质分类方案。 20)试述SWISS-PROT中的数据来源。 21)TrEMBL哪两个部分? 22)试述PSI-BLAST 搜索的5个步骤。 三、操作与计算题 1)如何获取访问号为U49845的genbank文件?解释如下genbank文件的LOCUS行提供的信息: LOCUS SCU49845 5028 bp DNA linear PLN 21-JUN-1999 2)利用Entrez检索系统,对核酸数据搜索,输入如下信息,将获得什
生物信息学的主要研究内容
常用数据库 在DNA序列方面有GenBank、EMBL和等 在蛋白质一级结构方面有SWISS-PROT、PIR和MIPS等 在蛋白质和其它生物大分子的结构方面有PDB等 在蛋白质结构分类方面有SCOP和CATH等 生物信息学的主要研究内容 1、序列比对(Alignment) 基本问题是比较两个或两个以上符号序列的相似性或不相似性。序列比对是生物信息学的基础,非常重要。两个序列的比对有较成熟的动态规划算法,以及在此基础上编写的比对软件包BLAST和FASTA,可以免费下载使用。这些软件在数据库查询和搜索中有重要的应用。 2、结构比对 基本问题是比较两个或两个以上蛋白质分子空间结构的相似性或不相似性。已有一些算法。 3、蛋白质结构预测,包括2级和3级结构预测,是最重要的课题之一 从方法上来看有演绎法和归纳法两种途径。前者主要是从一些基本原理或假设出发来预测和研究蛋白质的结构和折叠过程。分子力学和分子动力学属这一范畴。后者主要是从观察和总结已知结构的蛋白质结构规律出发来预测未知蛋白质的结构。同源模建(Homology)和指认(Threading)方法属于这一范畴。虽然经过30余年的努力,蛋白结构预测研究现状远远不能满足实际需要。 4、计算机辅助基因识别(仅指蛋白质编码基因)。最重要的课题之一 基本问题是给定基因组序列后,正确识别基因的范围和在基因组序列中的精确位置.这是最重要的课题之一,而且越来越重要。经过20余年的努力,提出了数十种算法,有十种左右重要的算法和相应软件上网提供免费服务。原核生物计算机辅助基因识别相对容易些,结果好一些。从具有较多内含子的真核生物基因组序列中正确识别出起始密码子、剪切位点和终止密码子,是个相当困难的问题,研究现状不能令人满意,仍有大量的工作要做。 5、非编码区分析和DNA语言研究,是最重要的课题之一 在人类基因组中,编码部分进展总序列的3~5%,其它通常称为“垃圾”DNA,其实一点也不是垃圾,只是我们暂时还不知道其重要的功能。分析非编码区DNA 序列需要大胆的想象和崭新的研究思路和方法。DNA序列作为一种遗传语言,不仅体现在编码序列之中,而且隐含在非编码序列之中。 6、分子进化和比较基因组学,是最重要的课题之一 早期的工作主要是利用不同物种中同一种基因序列的异同来研究生物的进化,构建进化树。既可以用DNA序列也可以用其编码的氨基酸序列来做,甚至于可通过相关蛋白质的结构比对来研究分子进化。以上研究已经积累了大量的工作。近年来由于较多模式生物基因组测序任务的完成,为从整个基因组的角度来研究分子进化提供了条件。 7、序列重叠群(Contigs)装配 一般来说,根据现行的测序技术,每次反应只能测出500或更多一些碱基对的序列,这就有一个把大量的较短的序列全体构成了重叠群(Contigs)。逐步把它们拼接起来形成序列更长的重叠群,直至得到完整序列的过程称为重叠群装配。拼接EST数据以发现全长新基因也有类似的问题。已经证明,这是一个NP-完备
网上生物信息学教程
网上生物信息学教程EMBL biocomputing tutorials https://www.wendangku.net/doc/645065839.html,/Embnetut/Gcg/index.html Plant genome dababase tutorial https://www.wendangku.net/doc/645065839.html,/pgdic 生物信息学机 NCBI https://www.wendangku.net/doc/645065839.html,/ International Nucleotide Sequence Database Collaboration. https://www.wendangku.net/doc/645065839.html,/collab/ EBI https://www.wendangku.net/doc/645065839.html,/ USDA https://www.wendangku.net/doc/645065839.html,/ Sanger Centre https://www.wendangku.net/doc/645065839.html,/ 北京大学生物信息学中心 https://www.wendangku.net/doc/645065839.html, 数据库信息发布及其它 GenBank Release Notes ftp://https://www.wendangku.net/doc/645065839.html,/genbank/gbrel.txt dbEST summary report https://www.wendangku.net/doc/645065839.html,/dbEST/dbESTsummarv.html EMBL release notes http://www.bio.unizh.ch/db/docu.html?data=emrel Eukaryotic promoter database release notes http://www.genome.ad.jp/dbget/dbget2.html KEGG release notes http://www.genome.jp/kegg/docs/relnote.html 核苷酸数据库 GenBank https://www.wendangku.net/doc/645065839.html,/ dbEST https://www.wendangku.net/doc/645065839.html,/dbEST/index.html dbSTS https://www.wendangku.net/doc/645065839.html,/dbSTS/index.html dbGSS https://www.wendangku.net/doc/645065839.html,/dbGSS/index.html
最新生物信息学名词解释(个人整理)
一、名词解释: 1.生物信息学:研究大量生物数据复杂关系的学科,其特征是多学科交叉,以互联网为媒介,数据库为载体。利用数学知识建立各种数学模型; 利用计算机为工具对实验所得大量生物学数据进行储存、检索、处理及分析,并以生物学知识对结果进行解释。 2.二级数据库:在一级数据库、实验数据和理论分析的基础上针对特定目标衍生而来,是对生物学知识和信息的进一步的整理。 3.FASTA序列格式:是将DNA或者蛋白质序列表示为一个带有一些标记的核苷酸或者氨基酸字符串,大于号(>)表示一个新文件的开始,其他无特殊要求。 4.genbank序列格式:是GenBank 数据库的基本信息单位,是最为广泛的生物信息学序列格式之一。该文件格式按域划分为4个部分:第一部分包含整个记录的信息(描述符);第二部分包含注释;第三部分是引文区,提供了这个记录的科学依据;第四部分是核苷酸序列本身,以“//”结尾。 5.Entrez检索系统:是NCBI开发的核心检索系统,集成了NCBI的各种数据库,具有链接的数据库多,使用方便,能够进行交叉索引等特点。 6.BLAST:基本局部比对搜索工具,用于相似性搜索的工具,对需要进行检索的序列与数据库中的每个序列做相似性比较。P94 7.查询序列(query sequence):也称被检索序列,用来在数据库中检索并进行相似性比较的序列。P98 8.打分矩阵(scoring matrix):在相似性检索中对序列两两比对的质量评估方法。包括基于理论(如考虑核酸和氨基酸之间的类似性)和实际进化距离(如PAM)两类方法。P29 9.空位(gap):在序列比对时,由于序列长度不同,需要插入一个或几个位点以取得最佳比对结果,这样在其中一序列上产生中断现象,这些中断的位点称为空位。P29 10.空位罚分:空位罚分是为了补偿插入和缺失对序列相似性的影响,序列中的空位的引入不代表真正的进化事件,所以要对其进行罚分,空位罚分的多少直接影响对比的结果。P37 11.E值:衡量序列之间相似性是否显著的期望值。E值大小说明了可以找到与查询序列(query)相匹配的随机或无关序列的概率,E值越接近零,越不可能找到其他匹配序列,E 值越小意味着序列的相似性偶然发生的机会越小,也即相似性越能反映真实的生物学意义。P95 12.低复杂度区域:BLAST搜索的过滤选项。指序列中包含的重复度高的区域,如poly(A)。 13.点矩阵(dot matrix):构建一个二维矩阵,其X轴是一条序列,Y轴是另一个序列,然后在2个序列相同碱基的对应位置(x,y)加点,如果两条序列完全相同则会形成一条主对角线,如果两条序列相似则会出现一条或者几条直线;如果完全没有相似性则不能连成直线。 14.多序列比对:通过序列的相似性检索得到许多相似性序列,将这些序列做一个总体的比对,以观察它们在结构上的异同,来回答大量的生物学问题。 15.分子钟:认为分子进化速率是恒定的或者几乎恒定的假说,从而可以通过分子进化推断出物种起源的时间。 16.系统发育分析:通过一组相关的基因或者蛋白质的多序列比对或其他性状,可以研究推断不同物种或基因之间的进化关系。 17.进化树的二歧分叉结构:指在进化树上任何一个分支节点,一个父分支都只能被分成两个子分支。 系统发育图:用枝长表示进化时间的系统树称为系统发育图,是引入时间概念的支序图。 18.直系同源:指由于物种形成事件来自一个共同祖先的不同物种中的同源序列,具有相似或不同的功能。(书:在缺乏任何基因复制证据的情况下,具有共同祖先和相同功能的同源基因。)
生物信息学复习资料全
一、名词解释(31个) 1.生物信息学:广义:应用信息科学的方法和技术,研究生物体系和生物过程 息的存贮、信息的涵和信息的传递,研究和分析生物体细胞、组织、器官的生理、病理、药理过程中的各种生物信息,或者也可以说成是生命科学中的信息科学。狭义:应用信息科学的理论、方法和技术,管理、分析和利用生物分子数据。 2.二级数据库:对原始生物分子数据进行整理、分类的结果,是在一级数据库、 实验数据和理论分析的基础上针对特定的应用目标而建立的。 3.多序列比对:研究的是多个序列的共性。序列的多重比对可用来搜索基因组 序列的功能区域,也可用于研究一组蛋白质之间的进化关系。 4.系统发育分析:是研究物种进化和系统分类的一种方法,其常用一种类似树 状分支的图形来概括各种(类)生物之间的亲缘关系,这种树状分支的图形称为系统发育树。 5.直系同源:如果由于进化压力来维持特定模体的话,模体中的组成蛋白应该 是进化保守的并且在其他物种中具有直系同源性。 指的是不同物种之间的同源性,例如蛋白质的同源性,DNA序列的同源性。(来自百度) 6.旁系(并系)同源:是那些在一定物种中的来源于基因复制的蛋白,可能会 进化出新的与原来有关的功能。用来描述在同一物种由于基因复制而分离的同源基因。(来自百度) 7.FASTA序列格式:将一个DNA或者蛋白质序列表示为一个带有一些标记的 核苷酸或氨基酸字符串。 8.开放阅读框(ORF):是结构基因的正常核苷酸序列,从起始密码子到终止 密码子的阅读框可编码完整的多肽链,其间不存在使翻译中断的终止密码子。(来自百度) 9.结构域:大分子蛋白质的三级结构常可分割成一个或数个球状或纤维状的区 域,折叠得较为紧密,各行其功能,称为结构域。 10.空位罚分:序列比对分析时为了反映核酸或氨基酸的插入或缺失等而插入空 位并进行罚分,以控制空位插入的合理性。(来自百度) 11.表达序列标签:通过从cDNA文库中随机挑选的克隆进行测序所获得的部分 cDNA的3’或5’端序列。(来自文献) 12.Gene Ontology 协会: 13.HMM 隐马尔可夫模型:将核苷酸序列看成一个随机序列,DNA序列的编 码部分与非编码部分在核苷酸的选用频率上对应着不同的Markov模型。14.一级数据库:数据库中的数据直接来源于实验获得的原始数据,只经过简单 的归类整理和注释 15.序列一致性:指同源DNA顺序的同一碱基位置的相同的碱基成员, 或者蛋 白质的同一氨基酸位置的相同的氨基酸成员, 可用百分比表示。 16.序列相似性:指同源蛋白质的氨基酸序列中一致性氨基酸和可取代氨基酸所 占的比例。 17.Blastn:是核酸序列到核酸库中的一种查询。库中存在的每条已知序列都将 同所查序列作一对一地核酸序列比对。(来自百度) 18.Blastp:是蛋白序列到蛋白库中的一种查询。库中存在的每条已知序列将逐 一地同每条所查序列作一对一的序列比对。(来自百度)
最新生物信息学学习心得
生物信息学学习心得 第一篇:生物信息学 生物信息学是上世纪90年代初人类基因组计划(hgp)依赖,随着基因组学、蛋白组学等新兴学科的建立,逐渐发展起来的生物学、数学和计算机信息科学的一门交叉应用学科。目前生物信息学的研究领域主要包括基于生物序列数据的整理和注释、生物信息挖掘工具开发及利用这些工具揭示生物学基础理论知识等领域。生物信息学作为新型交叉应用学科,可以依托本校已有的计算机科学、信息学、生物学和数学等学科优势,充分展现投入少、见效快、起点高的特色,推动学校学科建设和本科教学水平。 本实验指导书中的8个实验均设计为综合性开发实验,面向生物信息学院全体本科学生和研究生,以及全校对生物信息学感兴趣的其他专业学生开放。生物信息学实验室将提供系统的保障,包括采用mail服务器和linux帐号管理等进行实验过程管理和支持。限选《生物信息学及实验》的生物技术专业本科生至少选择其中5个实验,并不少于8个学时,即为课程要求的0.5个学分。其他选修者按照课时和学校相关规定计算创新学分。实验一熟悉生物信息学网站及其数据的生物学意义 实验目的:
培养学生利用互联网资源获取生物信息学研究前沿和相关数据的能力,熟悉生物信息学相关的一些重要国内外网站,及其核酸序列、蛋白质序列及代谢途径等功能相关数据库,学会下载生物相关的信息数据,了解不同的数据文件格式和其中重要的生物学意义。 实验原理: 利用互联网资源检索相关的国内外生物信息学相关网站,如:ncbi、sanger、tigr、kegg、sble、中科院北京基因组研究所、北大生物信息 学中心等,下载其中相关的数据,如fasta、genbank格式的核算和蛋白质序列、pathatdb格式化库文件,并输入blast命令进行计算,获得结果文件。 实验内容: 1. 向网上blast服务器提交序列,得到匹配结果; 2. 本地使用blast,格式化库文件,输入命令行得到匹配结果;
生物信息学名词解释(0001)
生物信息学名词解释
1.生物信息学:研究大量生物数据复杂关系的学科,其特征是多学科交叉,以互联网为媒介,数据库为载体。利用数学知识建立各种数学模型; 利用计算机为工具对实验所得大量生物学数据进行储存、检索、处理及分析,并以生物学知识对结果进行解释。 2.二级数据库:在一级数据库、实验数据和理论分析的基础上针对特定目标衍生而来,是对生物学知识和信息的进一步的整理。 3.FASTA序列格式:是将DNA或者蛋白质序列表示为一个带有一些标记的核苷酸或者氨基酸字符串,大于号(>)表示一个新文件的开始,其他无特殊要求。 4.genbank序列格式:是GenBank 数据库的基本信息单位,是最为广泛的生物信息学序列格式之一。该文件格式按域划分为4个部分:第一部分包含整个记录的信息(描述符);第二部分包含注释;第三部分是引文区,提供了这个记录的科学依据;第四部分是核苷酸序列本身,以“//”结尾。 5.Entrez检索系统:是NCBI开发的核心检索系统,集成了NCBI 的各种数据库,具有链接的数据库多,使用方便,能够进行交叉索引等特点。 6.BLAST:基本局部比对搜索工具,用于相似性搜索的工具,对需要进行检索的序列与数据库中的每个序列做相似性比较。P94 7.查询序列(query sequence):也称被检索序列,用来在数据库中检索并进行相似性比较的序列。P98 8.打分矩阵(scoring matrix):在相似性检索中对序列两两比对的质量评估方法。包括基于理论(如考虑核酸和氨基酸之间的类似性)和实际进化距离(如PAM)两类方法。P29 9.空位(gap):在序列比对时,由于序列长度不同,需要插入一个或几个位点以取得最佳比对结果,这样在其中一序列上产生中断现象,这些中断的位点称为空位。P29 10.空位罚分:空位罚分是为了补偿插入和缺失对序列相似性的影 响,序列中的空位的引入不代表真正的进化事件,所以要对其进行 罚分,空位罚分的多少直接影响对比的结果。P37 11.E值:衡量序列之间相似性是否显著的期望值。E值大小说明了 可以找到与查询序列(query)相匹配的随机或无关序列的概率,E 值越接近零,越不可能找到其他匹配序列,E值越小意味着序列的 相似性偶然发生的机会越小,也即相似性越能反映真实的生物学意 义。P95 12.低复杂度区域:BLAST搜索的过滤选项。指序列中包含的重复 度高的区域,如poly(A)。 13.点矩阵(dot matrix):构建一个二维矩阵,其X轴是一条序列, Y轴是另一个序列,然后在2个序列相同碱基的对应位置(x,y) 加点,如果两条序列完全相同则会形成一条主对角线,如果两条序 列相似则会出现一条或者几条直线;如果完全没有相似性则不能连 成直线。 14.多序列比对:通过序列的相似性检索得到许多相似性序列,将这 些序列做一个总体的比对,以观察它们在结构上的异同,来回答大 量的生物学问题。 15.分子钟:认为分子进化速率是恒定的或者几乎恒定的假说,从而 可以通过分子进化推断出物种起源的时间。 16.系统发育分析:通过一组相关的基因或者蛋白质的多序列比对或 其他性状,可以研究推断不同物种或基因之间的进化关系。 17.进化树的二歧分叉结构:指在进化树上任何一个分支节点,一个 父分支都只能被分成两个子分支。 系统发育图:用枝长表示进化时间的系统树称为系统发育图,是 引入时间概念的支序图。 18.直系同源:指由于物种形成事件来自一个共同祖先的不同物种中 的同源序列,具有相似或不同的功能。(书:在缺乏任何基因复制 证据的情况下,具有共同祖先和相同功能的同源基因。) 19.旁系(并系)同源:指同一个物种中具有共同祖先,通过基因重 复产生的一组基因,这些基因在功能上可能发生了改变。(书:由于 基因重复事件产生的相似序列。) 20.外类群:是进化树中处于一组被分析物种之外的,具有相近亲缘 关系的物种。 21.有根树:能够确定所有分析物种的共同祖先的进化树。 22.除权配对算法(UPGMA):最初,每个序列归为一类,然后找 到距离最近的两类将其归为一类,定义为一个节点,重复这个过程, 直到所有的聚类被加入,最终产生树根。 23.邻接法(neighbor-joining method):是一种不仅仅计算两两比 对距离,还对整个树的长度进行最小化,从而对树的拓扑结构进行 限制,能够克服UPGMA算法要求进化速率保持恒定的缺陷。 24.最大简约法(MP):在一系列能够解释序列差异的的进化树中 找到具有最少核酸或氨基酸替换的进化树。 25.最大似然法(ML):它对每个可能的进化位点分配一个概率, 然后综合所有位点,找到概率最大的进化树。最大似然法允许采用 不同的进化模型对变异进行分析评估,并在此基础上构建系统发育 树。 26.一致树(consensus tree):在同一算法中产生多个最优树,合并 这些最优树得到的树即一致树。 27.自举法检验(Bootstrap):放回式抽样统计法。通过对数据集多 次重复取样,构建多个进化树,用来检查给定树的分枝可信度。
生物信息学课程大纲
《生物信息学》课程大纲 一、课程目标 1.学科素养:生物信息学的基本研究方法,并能掌握应用其中的一些常用方法,以提高学生的科研能力,领会采用信息学技术去分析和探索大量核酸和蛋白质序列所蕴藏的生命意义的基本思路。(支撑毕业要求 3-1) 2.交流合作:理解团队学习的重要性,具有团队协作精神,掌握沟通合作技能,具有小组互助和协作学习体验。(支撑毕业要求 8-1) 3.交流合作:具有小组互助和协作学习体验,具备与学校领导、同事、学生、家长及社区沟通交流的知识与技能。(支撑毕业要求 8-3)
二、课程目标与毕业要求的对应关系 三、教学内容、重难点和课时安排 1.第一章生物信息学概论(2学时) (一)教学要求 理解和掌握相关名词的定义、概念和解释等,形成正确的理念和行为观点(二)教学内容 1、生物信息学的概念和发展历史(支撑课程目标1) 2、生物信息学的生物学基础(支撑课程目标1) 3、生物信息学的计算机和网络基础(支撑课程目标1) 4、生物信息学的数学基础(支撑课程目标1) 5、生物信息学的产业化(支撑课程目标1) 6、生物信息学研究内容和发展前景展望(支撑课程目标1)
(三)本章重难点 ※生物信息学的数学基础、生物信息学的计算机和网络基础 第二章分子生物学数据库(2学时) (一)教学要求 理解和掌握相关名词的定义、概念和解释等,形成正确的理念和行为观点 (二)教学内容 1、生物学数据库概述(支撑课程目标1) 2、核苷酸序列与基因组数据库(支撑课程目标1) 3、蛋白质序列与模式、同源性数据库(支撑课程目标1) 4、结构数据库(支撑课程目标1) 5、基因和分子的互作和代谢途径信息数据库(支撑课程目标1) 6、RNA核苷酸序列数据库(支撑课程目标1) 7、其它遗传学与分子生物学资源(支撑课程目标1) 8、数据库中存在的问题及使用注意事项(支撑课程目标1) (三)本章重难点 ※蛋白质序列与模式、同源性数据库;基因和分子的互作和代谢途径信息数据库;RNA 核苷酸序列数据库 第三章序列比对与数据库检索(2学时) (一)教学要求 理解和掌握相关名词的定义、概念和解释等,形成正确的理念和行为观点 (二)教学内容 1、序列比对概述(支撑课程目标1) 2、双序列比对(支撑课程目标1) 3、比对的统计学显著性(支撑课程目标1) 4、多序列比对(支撑课程目标1) 5、数据库搜索(支撑课程目标1) 6、基因组长序列比对(支撑课程目标1) (三)本章重难点 ※双序列比对、数据库搜索 第四章核酸序列的信号和功能识别(4学时)
2021高中生物最新辅导书籍推荐
2021高中生物最新辅导书籍推荐 许多准备参加生物竞赛的高中小盆友总会向别人请教买什么辅导资料最好,问的人多了,答案也就五花八门,然并卵,在这浩如烟海的书目中也是茫然无措。特此归纳整理了各方建议,希望能对生竞选手们有所帮助。 首先,要知道什么是生物联赛。我们通常所说的中学生物学奥赛是分为以下五个赛程的:各省的初赛、全国中学生生物学联赛、全国中学生生物学竞赛、全国中学生生物学冬令营、国际中学生生物学奥林匹克竞赛即IBO。就是通过这层层的严格选拔,在全国范围内发掘出高手中的高手作为国家对选手参加IBO,为国争光。而当下由于很多高校都把学科竞赛省赛成绩作为自主招生申请条件之一,所以作为第二阶段的全国中学生生物学联赛也就获得更多的关注了。 其次,要知道生物联赛的考核内容。我们都知道该考试以高中生物学为基础,并会扩展至高校普通生物学内容,具体考核点与分值分布是这样的: 1.细胞生物学、生物化学、微生物学、生物信息学 25% 2.植物和动物的解剖、生理、组织和器官的结构与功能 30% 3.动物行为学、生态学 20% 4.遗传学与进化生物学、生物系统学 25% 最后回归主题吧,到底该准备些什么备考资料呢? 入门篇: 吴相钰著《陈阅增普通生物学》——高等教育出版社看过之后对生物有个大致的概念 尹长明著《生物奥林匹克竞赛教程》-——湖南师范大学出版社 北京大学生物学家编著《精英教案》基础生物教程上、中、下册——军事谊文出版社 北京大学生物学家编著《精英教案》生物习题专集——军事谊文出版社 拔高篇: 刘凌云著《细胞生物学》——高等教育出版社 刘凌云、郑光美著《普通动物学》——高等教育出版社 王玢、左明雪著《人体及动物生理学》-——高等教育出版社
国内外生物信息学发展状况
国内外生物信息学发展状况 1.国外生物信息发展状况 国外非常重视生物信息学的发展各种专业研究机构和公司如雨后春笋般涌现出来,生物科技公司和制药工业内部的生物 信息学部门的数量也与日俱增。美国早在1988年在国会的支持 下就成立了国家生物技术信息中心(NCBI),其目的是进行计 算分子生物学的基础研究,构建和散布分子生物学数据库;欧 洲于1993年3月就着手建立欧洲生物信息学研究所(EBI), 日本也于1995年4月组建了信息生物学中心(CIB)。目前, 绝大部分的核酸和蛋白质数据库由美国、欧洲和日本的3家数 据库系统产生,他们共同组成了 DDBJ/EMBL/Gen Bank国际核 酸序列数据库,每天交换数据,同步更新。以西欧各国为主的 欧洲分子生物学网络组织(EuropeanMolecular Biology Network, EMB Net)是目前国际最大的分子生物信息研究、开 发和服务机构,通过计算机网络使英、德法、瑞士等国生物信 息资源实现共享。在共享网络资源的同时,他们又分别建有自 己的生物信息学机构、二级或更高级的具有各自特色的专业数 据库以及自己的分析技术,服务于本国生物(医学)研究和开 发,有些服务也开放于全世界。 从专业出版业来看,1970年,出现了《Computer Methods and Programs in Biomedicine》这本期刊;到1985年4月, 就有了第一种生物信息学专业期刊《Computer Application
in the Biosciences》。现在,我们可以看到的专业期刊已经很多了。 2 国内生物信息学发展状况 我国生物信息学研究近年来发展较快,相继成立了北京大学生物信息学中心、华大基因组信息学研究中心、中国科学院上海生命科学院生物信息中心,部分高校已经或准备开设生物信息学专业。2002年国家自然科学基金委在生物化学、生物物理学与生物医学工程学学科设立了生物信息学项目,并列入生命科学部优先资助的研究项目。国家 863计划特别设立了生物信息技术主题,从国家需求的层面上推动我国生物信息技术的大力发展[3]。 但是由于起步较晚及诸多原因,我国的生物信息学发展水平远远落后于国外。在PubMed收录的以关键词“Bioinformatics”检索到的历年发表的文章数,可以看出大量的研究文献出现在21世纪以后。其中我国共有138篇占全部5548篇的2.5%,而美国则发表2160篇占全部的39%之多(统计数据截至2004年2月15日)。我国学者在生物信息学领域发表的有高影响力的论文只有不到美国学者发表数量的6%,差距相当大[4]。在生物信息学领域,一些著名院士和教授在各自领域取得了一定成绩,显露出蓬勃发展的势头,有的在国际上还占有一席之地。如北京大学的罗静初和顾孝诚教授在生物信息学网站建设方面、中科院生物物理所的陈润生研究员在EST
