限制性内切酶使用

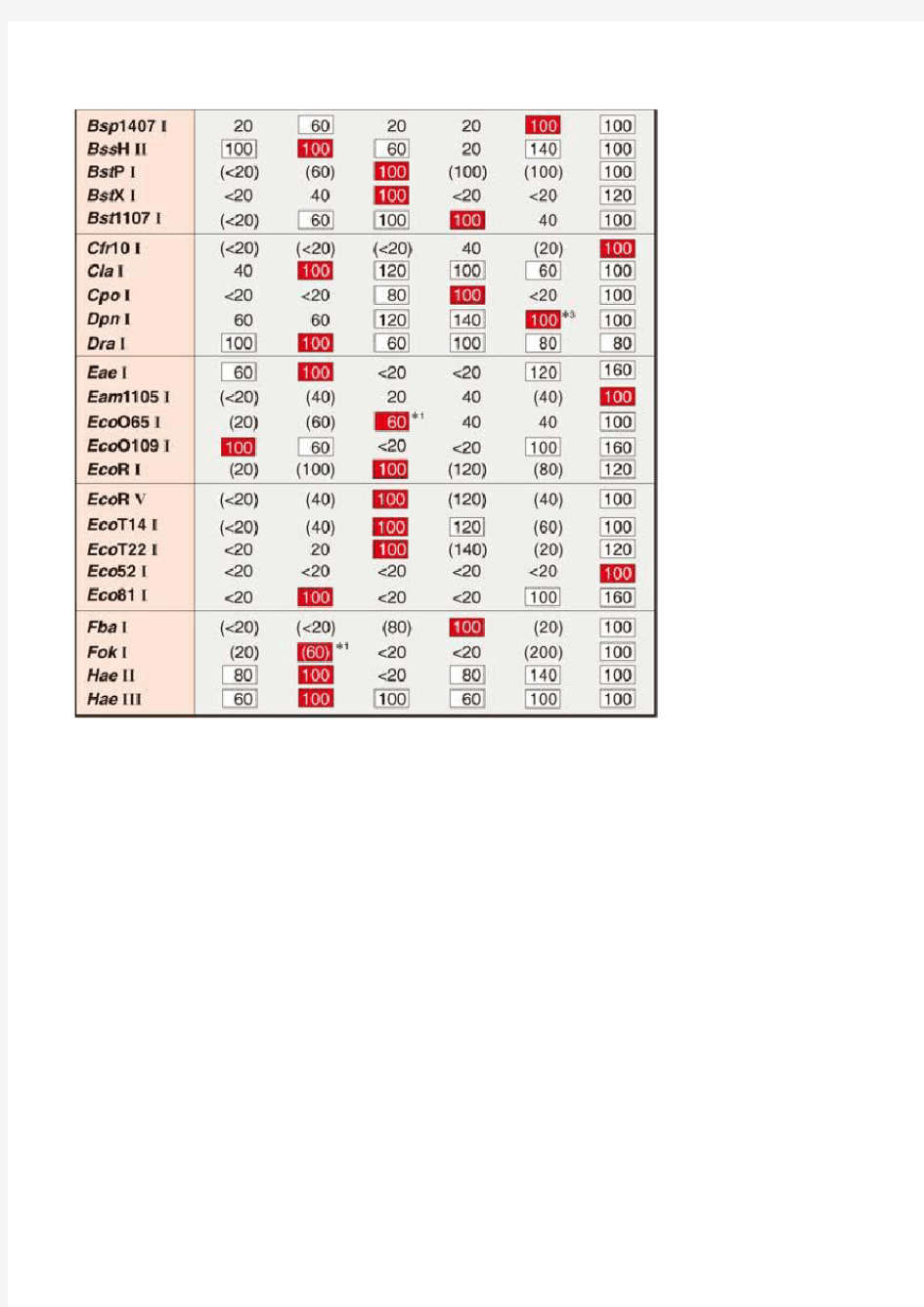
用Universal Buffer和Basal Buffer进行限制酶活性表示
TaKaRa公司,为了方便限制酶的统一使用,采用了通用缓冲液(Universal Buffer) 测定限制酶活性的体系(5种通用缓冲液中,用标注的),以此时的活性值作为100%。并把在其它通用缓冲液中的相对活性表示如下表。有( ) 标记的是易受Star活性影响的缓冲液,为了避免Star活性的影响,希望尽量使用或标注的缓冲液。每种限制酶都有其自身的基本缓冲液(Basal Buffer),其中AccⅢ、BalⅠ、BcnⅠ、BglⅠ、Bpu1102Ⅰ、Cfr10Ⅰ、Eam1105Ⅰ、Eco52Ⅰ、NruⅠ、Psh BⅠ、Sna BⅠ、Ssp Ⅰ、TaqⅠ、VpaK11B Ⅰ(共14种)由于没有十分合适的通用缓冲液,只能使用基本缓冲液(Basal Buffer)。各种限制酶的基本缓冲
液组成不同,相互之间不能通用。各种限制酶在基本缓冲液中的相对活性也被列于下表,供参考。
限制酶在各种缓冲液中的相对活性
附带·活性测定用Buffer 推荐使用的Buffer
*1+0.01%BSA→100%: Afl II, Aor13H I, Eco O65 I, Fok I, Hin1 I, Mun I, Nco I, Pvu I, Sse8387 I, Xba I *2 +0.01%BSA+0.01%Triton X-100→100%: Not I
按Universal Buffer分类的限制酶
各Universal Buffer的组成
■ 使用注意事项
10×Buffer都为10倍浓度的缓冲液。此外,10×T溶液中不含BSA,在使用时将BSA添加进去,使最终浓度为0.01%,有些限制酶(带有*1或*2标记)的反应体系中需加BSA或Triton X-100,添附的溶液是10倍浓度(0.1%) 的液体,使用时,请在反应体系中添加1/10量进行反应。
■ 保存
-20℃
反应停止液组份表
(10 × Loading Buffer)
1% SDS
60% Glycerol
0.05% Bromophenol Blue
■ 使用方法
本公司的酶包装中全部附带有反应停止液。使用时请添加反应液量的1/10,即可停止反应,进行电泳。-20℃保存时,会出现SDS 沉淀,请于温水浴中溶解后使用。在室温下保存时,SDS有时也会出现沉淀,此时同样请于温水浴中将其溶解后使用。
■ 保存
开封后室温保存。
Double Digestion(双酶切反应)时Universal Buffer(通用缓冲液)的使用表
■ 说明
使用二种酶同时进行DNA切断反应(Double Digestion) 时,为了节省反应时间,通常希望在同一反应体系内进行。TaKaRa采用Universal Buffer表示系统,并对每种酶表示了在各Universal Buffer中的相对活性。尽管如此,在进行Double Digestion时,有时还会难以找到合适的Universal Buffer。
本表以在pUC系列载体的多克隆位点处的各限制酶为核心,显示了在Double Digestion可使用的最佳Universal Buffer条件。在本表中,各Universal Buffer之前表示的[数字×] 是指各Universal Buffer的反应体系中的最终浓度。TaKaRa销售产品中添附的Universal Buffer全为10倍浓度的缓冲液。终浓度为0.5×时反应体系中的缓冲液则稀释至20倍,1×时稀释至10倍,2×时稀释至5倍进行使用。
■ 注意
◇ 1 μg DNA中添加10 U的限制酶,在50 μl的反应体系中,37℃下反应1小时可以完全降解DNA。
◇为防止Star活性的产生,请将反应体系中的甘油含量,尽量控制在10%以下。
◇根据DNA的种类,各DNA的立体结构的差别,或当限制酶识别位点邻接时,有时会发生Double Digestion不能顺利进行的可
能。
pUC18/19/118/119载体多克隆位点处的限制酶双酶切效果
在基因工程实验中,经常要对DNA片段进行多种限制酶的酶切反应。但经过实验验证,对在DNA片段上相邻或相近的两个限制酶切位点进行双酶切反应时,往往达不到预期的效果。例如,对pUC18/19/118/119载体多克隆位点处的Kpn I 和Sma I 进行双酶切时,酶切反应难以进行。而针对有些酶切位点进行双酶切时,酶切效果与酶切反应所用限制酶的顺序还有关系。例如,要对Sac I 和Kpn I 这两个酶切位点进行双酶切时,如果先用Sac I 进行酶切,然后再用Kpn I 进行酶切,双酶切反应则无法进行。但是如果先用Kpn I 进行酶切,然后再用Sac I 进行酶切,双酶切反应则可以进行。造成这些结果的原因是因为进行限制酶的双酶切反应时,由于受到酶切位点周围碱基数量的影响,从而影响了酶切效果。
本公司对pUC18/19/118/119载体多克隆位点处的限制酶进行双酶切时的效果,进行了实验验证,结果见表1和表2。表1是用两种限制酶进行分步酶切时的结果,表2是用两种限制酶进行同步酶切时的结果。
为了保证实验的顺利进行,对载体多克隆位点上的限制酶进行双酶切时,应该考虑各限制酶之间的位置关系。
下表中记号:“O”表示限制酶酶切效果较好;“△”表示限制酶的酶切效果不太好;“X”表示限制酶酶切效果很差。
表1 分步酶切效果
表2 同步酶切效果
注:
1. 对相邻酶切位点的限制酶进行双酶切反应时,同时加入限制酶进行双酶切反应的切断效果都不太理想,建议按先后顺序加入相应的限制酶进行酶切反应。
2. 使用EcoR I进行酶切反应时,酶切体系为20 μl,将酶的添加量控制在10 U左右,反应时间控制在2小时左右,双酶切效果较好。
PCR产物末端限制酶切位点的切断情况
克隆PCR产物的方法之一,是在PCR产物两端设计一定的限制酶切位点,经酶切后克隆至用相同酶切的载体中。但实验证明,大多数限制酶对裸露的酶切位点不能切断。必须在酶切位点旁边加上一个至几个保护碱基,才能使所定的限制酶对其识别位点进行有效切断。
下表列举了15种限制酶,分别比较了各种限制酶在其酶切位点旁边分别加0、1、2、3个保护碱基后的切断情况。表中的:(-) 为不能切断;(±) 为不能完全切断;(+) 为能完全切断。
结果显示,基本上所有限制酶,在其酶切位点旁边加上3个以上的保护碱基后,可以对其酶切位点进行有效切断。
常用限制性内切酶酶切位点汇总
Acc65I识别位点AccI识别位点AciI识别位点AclI识别位点AcuI识别位点 AfeI识别位点AflII识别位点AflIII识别位点AgeI识别位点AhdI识别位点AleI识别位点AluI识别位点AlwI识别位点AlwNI识别位点ApaI识别位点ApaLI识别位点ApeKI识别位点ApoI识别位点AscI识别位点AseI识别位点AsiSI识别位点AvaI识别位点AvaII识别位点AvrII识别位点BaeI识别位点BamHI识别位点BanI识别位点BanII识别位点
BbvCI识别位点BbvI识别位点 BccI识别位点BceAI识别位点BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点BmgBI识别位点BmrI识别位点BmtI识别位点BpmI识别位点Bpu10I识别位点BpuEI识别位点BsaAI识别位点BsaBI识别位点BsaHI识别位点BsaI识别位点BsaJI识别位点BsaWI识别位点BsaXI识别位点BseRI识别位点BseYI识别位点
BsiEI 识别位点BsiHKAI 识别位点BsiWI识别位点BslI 识别位点BsmAI识别位点 BsmBI识别位点BsmFI识别位点BsmI识别位点BsoBI识别位点Bsp1286I识别位点BspCNI识别位点BspDI识别位点BspEI识别位点BspHI识别位点BspMI识别位点BspQI识别位点BsrBI识别位点BsrDI识别位点BsrFI识别位点BsrGI识别位点BsrI识别位点BssHII识别位点BssKI识别位点BssSI识别位点BstAPI识别位点BstBI识别位点BstEII识别位点BstNI识别位点
限制性内切酶酶切位点汇总
Acc65I识别位点 AccI识别位点 AciI识别位点 AclI识别位点 AcuI识别位点 AfeI识别位点 AflII识别位点 AflIII识别位点 AgeI识别位点 AhdI识别位点 AleI识别位点 AluI识别位点 AlwI识别位点 AlwNI识别位点 ApaI识别位点 ApaLI识别位点 ApeKI识别位点 ApoI识别位点 AscI识别位点 AseI识别位点 AsiSI识别位点 AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI识别位点 BanI识别位点 BanII识别位点
BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点 BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点 BmgBI识别位点 BmrI识别位点 BmtI识别位点 BpmI识别位点 Bpu10I识别位点 BpuEI识别位点 BsaAI识别位点 BsaBI识别位点 BsaHI识别位点 BsaI识别位点 BsaJI识别位点 BsaWI识别位点 BsaXI识别位点 BseRI识别位点 BseYI识别位点
BsiEI识别位点 BsiHKAI识别位点 BsiWI识别位点 BslI识别位点 BsmAI识别位点 BsmBI识别位点 BsmFI识别位点 BsmI识别位点 BsoBI识别位点 Bsp1286I识别位点 BspCNI识别位点BspDI识别位点 BspEI识别位点 BspHI识别位点 BspMI识别位点 BspQI识别位点 BsrBI识别位点 BsrDI识别位点 BsrFI识别位点 BsrGI识别位点 BsrI识别位点 BssHII识别位点 BssKI识别位点 BssSI识别位点 BstAPI识别位点 BstBI识别位点 BstEII识别位点 BstNI识别位点
常用限制性内切酶酶切位点保护残基
酶切位点保护碱基-PCR引物设计用于限制性内切酶 发布: 2010-05-24 20:19| 来源:生物吧| 编辑:刘浩| 查看: 161 次 本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,KpnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,SacII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI, 为什么要添加保护碱基? 在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。 其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。 该如何添加保护碱基? 添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。 添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。 为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。 实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。取1μg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。反应缓冲液含70mM Tris-HCl (pH 7.6), 10 mM MgCl2, 5 mMDTT及适量的NaCl或KCl(视酶的具体要求而定)。20%的PAGE(7M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。 本实验采用自连接的寡核苷酸作为对照。若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
限制性核酸内切酶
限制性核酸内切酶 限制性核酸内切酶( restriction endonucleases ),简称限制酶,是一类能识别和切割双链 DNA 分子中的某些特定核苷酸序列的核酸水解酶,主要从细菌中分离得到。根据结构和功能特性,把限制酶分为Ⅰ、Ⅱ和Ⅲ型。Ⅰ型限制酶的切点不固定,很难形成稳定的、特异性切割末端;Ⅲ型限制酶对 DNA 链的识别序列是非对称的,不产生特异性的 DNA 片段,故基因工程实验中基本不用Ⅰ型和Ⅲ型限制酶。 Ⅱ型限制酶的主要作用是切割 DNA 分子,在 DNA 重组、构建新质粒、建立 DNA 的限制性酶切图谱、 DNA 的分子杂交、制备 DNA 的放射性探针、构建基因文库等方面起到重要作用,是基因工程重要的工具酶。 Ⅱ型限制性核酸内切酶的特点是:一般能识别和切割 4~8 个碱基对的核苷酸序列;大多数识别序列具有回文结构。 Ⅱ型限制性核酸内切酶的切割方式有三种:切割产生 5 ' 突出的粘性末端( sticky ends );切割产生 3 ' 突出的粘性末端;切割产生平头末端( blunt ends )。 Ⅰ型限制性内切酶既能催化宿主DNA的甲基化,又催化非甲基化的DNA的水解;而Ⅱ型限制性内切酶只催化非甲基化的DNA的水解。III型限制性内切酶同时具有修饰及认知切割的作用根据酶的功能特性、大小及反应时所需的辅助因子,限制性内切酶可分为两大类,即I类酶和Ⅱ酶。最早从大肠杆菌中发现的EcoK、EcoB就属于I类酶。反应过程中除需Mg2+外,还需要S-腺苷-L甲硫氨酸、ATP;在DNA分子上没有特异性的酶解片断,这是I、Ⅱ类酶之间最明显的差异。因此,I类酶作为DNA的分析工具价值不大。Ⅱ类酶有EcoR I、BamH I、Hind Ⅱ、Hind Ⅲ等。反应只需Mg2+;最重要的是在所识别的特定碱基顺序上有特异性的切点,因而DNA分子经过Ⅱ类酶作用后,可产生特异性的酶解片断,这些片断可用凝胶电泳法进行分离、鉴别。 限制酶一般不切割自身的DNA分子,只切割外源DNA。 每种限制酶特异识别专一DNA序列,并在切割位点将其准确切割。 限制酶是基因工程用来切割目的基因的酶,DNA复制不需要。 DNA复制需要的是解旋酶和DNA聚合酶。 根据限制酶的结构,辅因子的需求切位与作用方式,可将限制酶分为三种类型 第一型限制酶:同时具有修饰(modification)及认知切割(restriction)的作用;另有认知(recognize)DNA上特定碱基序列的能力,通常其切割位(cleavage site)距离认知位(recognition site)可达数千个碱基之远。例如:EcoB、EcoK。 第二型限制酶:只具有认知切割的作用,修饰作用由其他酵素进行。所认知的位置多为短的回文序列(palindrome sequence);所剪切的碱基序列通常即为所认知的序列。是遗传工程上,实用性较高的限制酶种类。例如:EcoRI、HindⅢ。 第三型限制酶:与第一型限制酶类似,同时具有修饰及认知切割的作用。可认知短的不对称序列,切割位与认知序列约距24-26个碱基对。例如:EcoPI、HinfⅢ。 甲基化(DNA methylation) DNA甲基化是最早发现的修饰途径之一,大量研究表明,DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。
常用限制性内切酶酶切位点
AatII 识别位点 Acc65I 识别位点 AccI 识别位点 AciI 识别位点 AclI 识别位点 AcuI 识别位点 AfeI 识别位点 AflII 识别位点 AflIII 识别位点 AgeI 识别位点 AhdI 识别位点 AleI 识别位点 AluI 识别位点 AlwI 识别位点 AlwNI 识别位点 ApaI 识别位点 ApaLI 识别位点 ApeKI 识别位点 ApoI 识别位点 AscI 识别位点 AseI 识别位点 AsiSI 识别位点
AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI 识别位点 BanI识别位点 BanII识别位点 BbsI识别位点 BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点BcgI识别位点BciVI识别位点BclI识别位点 BfaI识别位点BfuAI识别位点BglI识别位点BglII识别位点BlpI识别位点Bme1580I识别位点BmgBI识别位点BmrI识别位点
BmtI 识别位点 BpmI 识别位点 Bpu10I 识别位点 BpuEI 识别位点 BsaAI 识别位点 BsaBI 识别位点 BsaHI 识别位点 BsaI 识别位点 BsaJI 识别位点 BsaWI 识别位点 BsaXI 识别位点 BseRI 识别位点 BseYI 识别位点 BsgI 识别位点 BsiEI 识别位点 BsiHKAI 识别位点 BsiWI 识别位点 BslI 识别位点 BsmAI 识别位点 BsmBI 识别位点 BsmFI 识别位点 BsmI 识别位点
限制性核酸内切酶与核酸内切酶、外切酶
限制性核酸内切酶百科名片
其3′→5′外切酶活性使双链DNA分子产生出单链区,经过这种修饰的DNA 再配合使用Klenow酶,同时加进带放射性同位素的核苷酸,便可以制备特异性的放射性探针。 核酸内切酶 核酸内切酶(endonuclease)在核酸水解酶中,为可水解分子链内部磷酸二酯键生成寡核苷酸的酶,与核酸外切酶相对应。从对底物的特异性来看,可分为DNaseⅠ、DNaseⅡ等仅分解DNA的酶;脾脏RNase、RNaseT1等仅分解RNA的酶。如链孢霉(Neurospora)的核酸酶就是既分解DNA又分解RNA的酶。一般来说,大都不具碱基特异性,但也有诸如脾脏RNase、RNaseT1等或限制性内切酶那种能够识别并切断特定的碱基或碱基序列的酶。[1] 寡核苷酸,是一类只有20个以下碱基对的短链核苷酸的总称(包括脱氧核糖核 酸DNA或核糖核酸RNA内的核苷酸),寡核苷酸可以很容易地和它们的互补对链接,所以常用来作为探针确定DNA或RNA的结构,经常用于基因芯片、电泳、荧光原位杂交等过程中。 RNA聚合酶 科技名词定义 中文名称:RNA聚合酶 英文名称:RNA polymerase 定义1:以一条DNA链或RNA链为模板催化由核苷-5′-三磷酸合成RNA的酶。 所属学科:生物化学与分子生物学(一级学科);酶(二级学科) 定义2:以一条DNA链或RNA链为模板催化由核苷-5′-三磷酸合成RNA的酶。 所属学科:细胞生物学(一级学科);细胞遗传(二级学科)
定义3:以DNA或RNA为模板合成RNA的酶。 所属学科:遗传学(一级学科);分子遗传学(二级学科) 本内容由全国科学技术名词审定委员会审定公布 RNA聚合酶(RNA polymerase):以一条DNA链或RNA为模板催化由核苷-5′-三磷酸合成RNA的酶。是催化以DNA为模板(template)、三磷酸核糖核苷为底物、通过磷酸二酯键而聚合的合成RNA的酶。因为在细胞内与基因DNA的遗传信息转录为RNA有关,所以也称转录酶。 逆转录酶 科技名词定义 中文名称:逆转录酶 英文名称:reverse transcriptase 其他名称:依赖于RNA的DNA聚合酶(RNA-dependent DNA polymerase,RNA指导的DNA聚合酶 (RNA-directed DNA polymerase) 定义:编号:EC 2.7.7.49。以RNA为模板催化脱氧核苷-5′-三磷酸合成DNA的酶。在逆转录病毒及其他某些病毒中发现有此类酶。 所属学科:生物化学与分子生物学(一级学科);酶(二级学科) 本内容由全国科学技术名词审定委员会审定公布
高中生物论文解读限制性核酸内切酶应用的考点例析人教版
解读《限制性核酸内切酶应用的考点例析》 我们知道限制性核酸内切酶(限制酶)是指能识别DNA中特定碱基顺序,并在特定位点切割双链DNA的核酸内切酶。它在生物学中应用相当广泛,是基因工程中的工具酶,用来构建重组DNA分子,对于遗传性疾病的基因定位和基因诊断的研究也具有重要的应用价值。下面我们以问题的形式简要地了解它在这些方面的应用。 1。限制酶的特点 例1.下面哪项不具有限制酶识别序列的特征() A.GAATTC B.GGGGCCCC C.CTGCAG D.CTAAATC CTTAAG CCCCGGGG GACGTC GATTTAG 解析:限制酶识别的各种序具有回文对称的特点。所谓回文对称序列就是当以不同的方向分别阅读DNA的两条互补链时,DNA的两条链上的碱基序列相同。如A中的DNA分子,其中一条链从左向右阅读碱基序列是GAA TTC,另一条互补链从右向左阅读碱基序列也是GAATTC。 答案:D 例2.限制酶HindⅢ酶切DNA的识别序列是AAGCTT,限制酶HpaⅡ酶切DNA的识别序列是CCGG。假定DNA分子中A、T、G、C所含的比例相等,那么,限制酶HindⅢ酶切割双链DNA的概率是,酶切位点间的平均距离约kb(千碱基);限制酶HpaⅡ酶切割双链DNA的概率是,酶切位点间的平均距离约kb。 解析:因为限制酶识别序列具有回文对称序列的特点,这两个序列在相应的互补链上又会呈现,因此我们只需考虑DNA的一条链即可。六碱基长HindⅢ识别序列AAGCTT出现的概率是(1/4)6或1/4096,因此HindⅢ酶切位点之间的平均距离大约为4 kb。同样的道理,4碱基长的HpaⅡ酶识别序列CCGG出现的概率是(1/4)4或1/256,因此HpaⅡ酶切位点的平均距离大约为0.25 kb。 2.黏性末端与限制酶类型的关系 例3.用同一种限制酶处理会产生相同的黏性末端,但用不同的限制酶处理也可能产生相同的黏性末端。下列所示的四个黏性末端是由()种限制酶作用产生的。 解析:不同的限制酶的识别序列和切割位点不同。要判断题中的4个黏性末端是由几种限制酶作用下产生的,不光要看共有几种黏性末端,更重要的是要看作用产生这些黏性末端的限制酶的识别序列和切割位点是否相同。经过分析,题中4幅图所示的黏性末端应该分别是由4种限制酶作用产生的,这4种酶的识别序列及切割位点依次是:G↓AATTC,C↓AA TTG,G↓TTAAC,C↓TTAAG。 答案:4 3.限制酶图谱分析 例4.一线性DNA分子分别用限制酶HindⅢ和SmaⅠ消化,然后用这两种酶混合消化,得到如下片段: HindⅢ 2.5 kb,5.0 kb SmaⅠ 2.0 kb,5.5 kb HindⅢ和SmaⅠ 2.5 kb,3.0 kb,2.0 kb (1)画出此丝性DNA分子的限制酶图谱。 (2)两酶混合消化的片段再用限制酶EcoRⅠ消化,结果导致凝胶上3.0 kb的片段消失,产
(整理)限制性内切核酸酶
第三章限制性内切核酸酶 一、填空题 1. 严格地说限制性内切核酸酶(restriction endonuclease)是指已被证明是的酶。基因工程中把 那些具有识别的内切核酸酶统称为限制性内切核酸酶。 2.年Luria和Human在T偶数噬菌体对大肠杆菌感染实验中首次发现了细菌的现象。 3.1970年,Smith和Wilcox从流感嗜血杆菌中分离到一种限制酶,能够特异性的切割DNA, 这个酶后来被命名为,这是第一个分离到的Ⅱ类限制性内切核酸酶。 4.通过比较用不同组合的限制性内切核酸酶处理某一特定基因区域所得到的不同大小的片段,可 以构建显示该区域各限制性内切核酸酶切点相互位置的。 5.Ⅱ类限制性内切核酸酶分子量较小.一般在20~40kDa,通常由——亚基所组成。它们的作用底物 为双链DNA,极少数Ⅱ类酶也可作用于单链DNA,或DNA/RNA杂合双链;这类酶的专一性强,它不仅对酶切点邻近的两个碱基有严格要求,而且对更远的碱基也有要求,因此,Ⅱ类酶既具有专一性,也具有专一性,一般在识别序列内切割。切割的方式有,产生末端的DNA片段或的DNA片段。作用时需要——作辅助因子,但不需要和 6.完全的回文序列具有两个基本的特点,就是:和 7.Ⅱ类限制性内切核酸酶一般识别个碱基,也有识别多序列的限制性内切核酸酶;根据对限制性内切核酸酶识别序列的分析,限制性内切核酸酶识别序列具有倾向,即它们在识别序列中含量较高。 8.EcoK是I类限制性内切核酸酶,分子组成是_______ 分子量是300kDa.在这些亚基中,α亚基具有 作用;β亚基具有的活性;γ亚基的作用则是, 9.个体之间DNA限制性片段长度的差异叫 10.限制性内切核酸酶是按属名和种名相结合的原则命名的,第一个大写字母取自,第二、三两个字母取自,第四个字母则用表示。 11.限制性内切核酸酶AcyI识别的序列是5’-GRCGYG-3’,,其中R,Y 12.在酶切反应管加完各种反应物后,需要离心2秒钟,其目的是和 13.部分酶切可采取的措施有:(1)(2)(3)等。 14.第一个分离的限制性内切核酸酶是;而第一个用于构建重组体的限制性内切核酸酶是 15.限制性内切核酸酶BsuRI和HaeⅢ的来源不同,但识别的序列都是,它们属于。 16.由于DNA是由4种碱基组成的,所以任何限制性内切核酸酶的切割频率的理沦值应该是· 17.SalI和NotI都是哺乳动物中识别序列稀有的酶,在哺乳动物基因组的5kb片段中,找到NotI切点 的概率是。 18.部分酶切是指控制反应条件,使得酶在DNA序列上的识别位点只有部分得到切割,它的理论依据 是。 19.Ⅰ类限制酶识别DNA的位点和切割的DNA位点是不同的.切割位点的识别结合有两种模型,一种是,另一种是。 20.限制性内切核酸酶通常保存在浓度的甘油溶液中。 二、判断题 1.限制与修饰现象是宿主的一种保护体系,它足通过对外源DNA的修饰和对自身DNA的限制实现的。 2.限制性内切核酸酶在DNA中的识别/切割位点的二级/三级结构也影响酶切效率, 一般来说, 完全切割质粒或病毒DNA,要比切割线状DNA需要更多的酶,最高的需要20倍, 3.如果限制性内切核酸酶的识别位点位于DNA分子的末端,那么接近末端的程度也影响切割,如 HpaII和MboI要求识别序列之前至少有一个碱基对存在才能切割。 4.能够产生防御病毒侵染的限制性内切核酸酶的细菌,其本身的基因组中没有被该核酸酶识别的序列。 5.限制性图谱与限制性片段长度多态性(RFLP)图谱的最显著的区别在于前者是一个物理图谱而后者
限制性内切酶的一般原则和建议!
限制性内切酶的一般原则和建议! 1.如何做酶切反应? 该问题看似什么简单: DNA中加上酶,然后保温一段时间就可以了。但是在实际操作过程中,我们不断听到:切不动,装不上。问题在什么地方?能系列生产限制性内切酶的公司国际上,就那么几个,位列前 3 的是NEB, Fermentas, SibEnzyme。这些公司提供酶的品质一般都能得到保证。您可以怀疑酶的质量问题,但是更多的问题来源于模板是否合适酶切要求。下面几点对你的酶切是有帮助的。 1) 成功酶切的关键是准备好模板DNA。DNA样品中不能含有有机溶剂(会使酶变性或产生星号货性),不能含有干扰酶活性的污染物质,不能含有高浓度的EDTA (TE中的EDTA浓度较低,对Mg的浓度影响较小);同时要对DNA甲基化程度及其对酶切效率的影响要做到心中有数。 2) 选用合适的酶。根据酶切序列选用,特别注意选用甲基化对酶活性的干扰。 3) 正确使用和保存酶。酶需要保存在-20度的低温环境中,只是在需要用酶才从冰箱中取出来。运输和临时存放时需要将酶至于冰上。手拿酶管时不要接触酶管下步含酶的部分,移酶时尽可能用长TIP, 避免污染。用完后需要及时送回原处。注意:酶通常是最后加。所有4) 反应体积需要根据实验目的定,常规的酶切一般要维持在10-50ul,酶切鉴定10-20ul就可以了。 5) 模板浓度问题:浓度过高,溶液黏度过大,酶不能有效扩散,酶切效果不会好。浓度过低,也会影响酶活性。 6) 注意模板用量和反应体积的关系。对酶用量,模板用量,反应体积等要素的确定需要的是时间和经验的积累。 7) 酶切反应的各个组分加完后,需要用TIP小心混匀几次,short spin 一下就可以保温了。一般不能使用振荡器混匀。 8) 反应温度的选择。一般反应都用37度,但是 Sma I 的最适合温度是25度,37度时酶仍表现出活性,但是效率下降50%。部分从耐热菌制备的酶需要在37度以上的温度反应,如Taq I的最适温度为65度,37度保温,效率仅为前者的1/10。 9) 反应时间的选择。一般酶切鉴定30分钟就可以了。要完全酶切可以采用少量的酶长时间反应,或较高的酶量短时间处理都可以达到。在使用高酶量的时候需要注意甘油的最终浓度不要超过5%,也就是说10ul的体系,酶的用量不要超过1ul。 10) 是否和如何终止反应?酶切鉴定之类的实验不需要特殊处理。灭活的手段:加入高浓度的EDTA;65度或80度热处理20-30分钟;部分从高温菌纯化出来的内切酶由于最适的反应温度比较高,热处理灭活不一定完全,需要用苯酚/氯仿/乙醇方法纯化;电泳回收也是实验室常用除酶的手段。 2.如果遇到酶切不动或切不完全,该怎么办? 要回答这么问题常常需要了解酶活性单位是如何确定,我们多次接到这样的问题:1个单位的酶能在60分钟内切1ug的DNA,为什么我们的DNA那么少切那么长时间也不能切开或切完全?从下面几个因素去考虑: 1) 酶是否有活性:酶的活性单位通常是在60分钟酶切1ug lambda DNA或特定线状DNA所需要的酶量。鉴定酶的活性高低不是用您待切的DNA模板,也不是别的公司的酶来判定。因为不同公司酶可能是从不同系统中纯化的,虽然识别位点相同,但是酶的特性可能是有差异的。鉴定酶必须使用使用说明书上认定的酶活确定的方式,通常需要用lambda DAN做模板来判定。同时如果酶对甲基化敏感,还需要用Dcm-, Dam-的DNA.不排除由于运输或分装不当导致酶活性下降,这种情况是很少发生。我们公
常用限制性内切酶酶切位点汇总
ApaI识别位点Acc65I识别位点 ApaLI识别位点AccI识别位点 ApeKI识别位点AciI识别位点 ApoI识别位点AclI识别位点 AscI识别位点AcuI识别位点 AseI识别位点AfeI识别位点 AsiSI识别位点AflII识别位点 AvaI识别位点AflIII识别位点 AvaII识别位点AgeI识别位点 AvrII识别位点AhdI识别位点 BaeI识别位点AleI识别位点 BamHI识别位点AluI识别位点 BanI识别位点AlwI识别位点 BanII识别位点AlwNI识别位点
BmrI识别位点BbvCI识别位点 BmtI识别位点BbvI识别位点 BpmI识别位点BccI识别位点 Bpu10I识别位点BceAI识别位点 BpuEI识别位点BcgI识别位点 BsaAI识别位点BciVI识别位点 BsaBI识别位点BclI识别位点 BsaHI识别位点BfaI识别位点 BsaI识别位点BfuAI识别位点 BsaJI识别位点BglI识别位点 BsaWI识别位点BglII识别位点 BsaXI识别位点BlpI识别位点 BseRI识别位点Bme1580I识别位点 BseYI识别位点BmgBI识别位点
BspMI识别位点BsiEI识别位点 BspQI识别位点BsiHKAI识别位点 BsrBI识别位点BsiWI识别位点 BsrDI识别位点BslI识别位点 BsrFI识别位点BsmAI识别位点 BsrGI识别位点BsmBI识别位点 BsrI识别位点BsmFI识别位点 BssHII识别位点BsmI识别位点 BssKI识别位点BsoBI识别位点 BssSI识别位点Bsp1286I识别位点 BstAPI识别位点BspCNI识别位点 BstBI识别位点BspDI识别位点 BstEII识别位点BspEI识别位点 BstNI识别位点BspHI识别位点
限制性核酸内切酶
限制性核酸内切酶 限制性核酸内切酶:是识别DNA的特异序列,并在识别位点或其周围切割双链DNA的一 类内切酶。 限制性核酸内切酶的分类: 根据限制酶的结构,辅因子的需求切位与作用方式,可将限制酶分为三种类型,分别是第一 型(Type I)、第二型(Type II)及第三型(Type III)。 第一型限制酶 同时具有修饰(modification)及认知切割(restriction)的作用;另有认知(recognize)DNA上特定 碱基序列的能力,通常其切割位(cleavage site)距离认知位(recognition site)可达数千个碱基之远,并不能准确定位切割位点,所以并不常用。例如:EcoB、EcoK。 第二型限制酶 只具有认知切割的作用,修饰作用由其他酵素进行。所认知的位置多为短的回文序列(palindrome sequence);所剪切的碱基序列通常即为所认知的序列。是遗传工程上,实用性较 高的限制酶种类。例如:EcoRI、HindIII。 第三型限制酶 与第一型限制酶类似,同时具有修饰及认知切割的作用。可认知短的不对称序列,切割位与 认知序列约距24-26个碱基对,并不能准确定位切割位点,所以并不常用。例如: EcoPI、HinfIII。 限制酶在遗传学方面的应用: 1、在甚因工程方面 利用能产生“粘性末端”的限制酶, 进行DNA的体外重组, 是较为方便的, 只要用同一 限制酶处理不同来源的DNA, 由于所产生的水解片段具有相同的粘性末端, 可以彼此“粘合”,再经连接酶处理, 就成为重组DNA分子了. 目前, 基因工程上, 限制酶主要应用于以下两方面 ? (1)目的基因与载体的重组 细菌细胞中的限制酶能水解外源DNA , 因此必须通过适当的载体(质粒或噬菌体)的帮助 才能将外源DNA引人受体细胞并在其中增殖和表达。将供体DNA与载体用同样的限制酶处理, 使载体带上各种各样的外源DNA片断, 然后引人受体细菌细胞增殖, 菌细胞增殖, 再筛 选出所需的菌株, 便获得带有某一目的基因的繁殖系.用这种方法, 已成功地将酵母菌的咪哇
AgeI限制性内切酶使用说明书
H om e ?P roducts ?AgeI AgeI Product Information FAQs Protocols Other Tools & Resources Related Products Description Properties and Usage Quality Control This enzyme has transitioned to an improved new buffer system. Visit https://www.wendangku.net/doc/b413984568.html, for further details. The new and current Double Digest Finder and current Activity/Performance Chart for the CutSmart buffer system are available. The previous version of the Double Digest Finder , as well as the previous Version of Activity/Performance Chart that use the former buffer system, are still available for your convenience. AgeI has High Fidelity (HF) and RE-Mix Master Mix versions available. Isoschizomers Catalog # Size Concentration Price Qty R0552S 300 units 5,000 units/ml $67.001R0552L 1,500 units 5,000 units/ml $268.001 Categories: Restriction Endonucleases: A Applications: Restriction Enzyme Digestion Description AgeI has a High Fidelity v ersion AgeI-HF? (NEB #R3552). High Fidelity (HF ?) Restriction Enzymes hav e 100% activ ity in CutSmart ? Buffer; single-buffer simplicity means more straightforward and streamlined sample processing. HF enzymes also exhibit dramatically reduced star activ ity. HF enzymes are all Time-Sav er qualified and can therefore cut substrate DNA in 5-15 minutes with the flexibility to digest ov ernight without degradation to DNA. Engineered with performance in mind, HF restriction enzymes are fully activ e under a broader range of conditions, minimizing off-target products, while offering flexibility in experimental design. Product Source An E. coli strain that carries the AgeI gene from Ruegeria gelatinovora (ATCC 25655). Reagents Supplied The following reagents are supplied with this product: Properties and Usage Unit Definition One unit is defined as the amount of enzyme required to digest 1 μg of λ DNA in 1 hour at 37°C in a total reaction of 50 μl. NEBuffer 1.110X
常见限制性内切酶识别序列(酶切位点)
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine. ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3' 当然,上面总结的这些肯定不全,要查找更多内切酶的识别序列,你还可以选择下面几种方法: 1. 查你所使用的内切酶的公司的目录或者网站;NEB网站上提供的识别序列图表下载 2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;
DNA限制性内切酶——酶切Buffer组分及其活性
DNA限制性内切酶——酶切Buffer组分及其活性 TaKaRa公司,为了方便限制酶的统一使用,采用了通用缓冲液(Universal Buffer) 测定限制酶活性的体系(5种通用缓冲液中,用标注的),以此时的活性值作为100%。并把在其它通用缓冲液中的相对活性表示如下表。有( ) 标记的是易受Star活性影响的缓冲液,为了避免Star活性的影响,希望尽量使用或标注的缓冲液。每种限制酶都有其自身的基本缓冲液(Basal Buffer),其中AccⅢ、BalⅠ、BcnⅠ、BglⅠ、Bpu1102Ⅰ、Cfr10Ⅰ、Eam1105Ⅰ、Eco52Ⅰ、NruⅠ、Psh BⅠ、Sna BⅠ、SspⅠ、TaqⅠ、VpaK11B Ⅰ(共14种)由于没有十分合适的通用缓冲液,只能使用基本缓冲液(Basal Buffer)。各种限制酶的基本缓冲液组成不同,相互之间不能通用。各种限制酶在基本缓冲液中的相对活性也被列于下表,供参考。 限制酶在各种缓冲液中的相对活性 附带·活性测定用Buffer 推荐使用的Buffer
*1+0.01%BSA→100%:Afl II, Aor13H I, Eco O65 I, Fok I, Hin1 I, Mun I, Nco I, Pvu I, Sse8387 I, Xba I *2 +0.01%BSA+0.01%Triton X-100→100%:Not I *3不加BSA
按Universal Buffer分类的限制酶
各Universal Buffer的组成 ■ 使用注意事项 10×Buffer都为10倍浓度的缓冲液。此外,10×T溶液中不含BSA,在使用时将BSA添加进去,使最终浓度为0.01%,有些限制酶(带有*1或*2标记)的反应体系中需加BSA或Triton X-100,添附的溶液是10倍浓度(0.1%) 的液体,使用时,请在反应体系中添加1/10量进行反应。 ■ 保存 -20℃ 反应停止液组份表 (10 × Loading Buffer) 1%SDS 60%Glycerol 0.05%Bromophenol Blue ■ 使用方法 本公司的酶包装中全部附带有反应停止液。使用时请添加反应液量的1/10,即可停止反应,进行电泳。-20℃保存时,会出现SDS 沉淀,请于温水浴中溶解后使用。在室温下保存时,SDS有时也会出现沉淀,此时同样请于温水浴中将其溶解后使用。 ■ 保存 开封后室温保存。
常用限制性内切酶酶切位点总结
常用限制性内切酶酶切位点总结
————————————————————————————————作者:————————————————————————————————日期:
Acc65I识别位点 AccI识别位点 AciI识别位点 AclI识别位点 AcuI识别位点 AfeI识别位点 AflII识别位点 AflIII识别位点 AgeI识别位点 AhdI识别位点 AleI识别位点 AluI识别位点 AlwI识别位点 AlwNI识别位点 ApaI识别位点 ApaLI识别位点 ApeKI识别位点 ApoI识别位点 AscI识别位点 AseI识别位点 AsiSI识别位点 AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI识别位点 BanI识别位点 BanII识别位点
BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点 BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点 BmgBI识别位点 BmrI识别位点 BmtI识别位点 BpmI识别位点 Bpu10I识别位点 BpuEI识别位点 BsaAI识别位点 BsaBI识别位点 BsaHI识别位点 BsaI识别位点 BsaJI识别位点 BsaWI识别位点 BsaXI识别位点 BseRI识别位点 BseYI识别位点
BsiEI识别位点 BsiHKAI识别位点 BsiWI识别位点 BslI识别位点 BsmAI识别位点 BsmBI识别位点 BsmFI识别位点 BsmI识别位点 BsoBI识别位点 Bsp1286I识别位点 BspCNI识别位点BspDI识别位点 BspEI识别位点 BspHI识别位点 BspMI识别位点 BspQI识别位点 BsrBI识别位点 BsrDI识别位点 BsrFI识别位点 BsrGI识别位点 BsrI识别位点 BssHII识别位点 BssKI识别位点 BssSI识别位点 BstAPI识别位点 BstBI识别位点 BstEII识别位点 BstNI识别位点
常用限制性内切酶酶切位点汇总复习过程
常用限制性内切酶酶切位点汇总
Acc65I识别位点AccI识别位点AciI识别位点AclI识别位点AcuI识别位点AfeI识别位点AflII识别位点AflIII识别位点AgeI识别位点AhdI识别位点AleI识别位点AluI识别位点 AlwI识别位点AlwNI识别位点ApaI识别位点ApaLI识别位点ApeKI识别位点ApoI识别位点AscI识别位点AseI识别位点AsiSI识别位点AvaI识别位点AvaII识别位点AvrII识别位点BaeI识别位点BamHI识别位点BanI识别位点BanII识别位点
BbvCI识别位点BbvI识别位点 BccI识别位点BceAI识别位点BcgI识别位点BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点BmgBI识别位点BmrI识别位点BmtI识别位点BpmI识别位点Bpu10I识别位点BpuEI识别位点BsaAI识别位点BsaBI识别位点BsaHI识别位点BsaI识别位点BsaJI识别位点BsaWI识别位点BsaXI识别位点BseRI识别位点BseYI识别位点
BsiEI识别位点BsiHKAI识别位点BsiWI识别位点BslI识别位点BsmAI识别位点BsmBI识别位点 BsmFI识别位点BsmI识别位点BsoBI识别位点Bsp1286I识别位点BspCNI识别位点BspDI识别位点 BspEI识别位点BspHI识别位点BspMI识别位点BspQI识别位点BsrBI识别位点BsrDI识别位点BsrFI识别位点BsrGI识别位点BsrI识别位点BssHII识别位点BssKI识别位点BssSI识别位点BstAPI识别位点BstBI识别位点BstEII识别位点BstNI识别位点
- 限制性内切酶和引物
- 常用限制性内切酶酶切位点
- 常用的限制性内切酶
- 常见限制性内切酶酶切位点
- 第一节工具酶限制性内切酶
- ((完整版))常用限制性内切酶酶切位点汇总,推荐文档
- 常用限制性内切酶酶切位点总结
- 常用限制性内切酶酶切位点汇总
- 限制性内切酶的一般原则和建议
- 常用限制性内切酶酶切位点保护残基
- 第一节 工具酶(限制性内切酶)
- 常见限制性内切酶识别序列(酶切位点)
- 基因工程中常用的工具酶
- 常用限制性内切酶酶切位点保护残基
- 常见分子实验限制性内切酶酶切位点大全
- 限制性内切酶的一般原则和建议!
- 常用限制性内切酶酶切位点汇总
- 常用限制性内切酶酶切位点汇总
- 限制性内切酶酶切位点汇总
- 常用限制性内切酶酶切位点
